El bacteriófago Phi-X174 y los extraterrestres
El bacteriófago Phi- X174
En 1977 el grupo de investigación de Fred Sanger publicó la secuencia del genoma completo del bacteriófago Phi-X174*, el cual posee un genoma circular de DNA de tan solo 5,386 bases. En este minúsculo genoma se codifican 11 genes. Lo más sorprendente es que 8 de ellos están empalmados en por lo menos una base (Figura 1). En particular, los genes E, K, A* y B están codificados en su totalidad en empalme con otros genes.
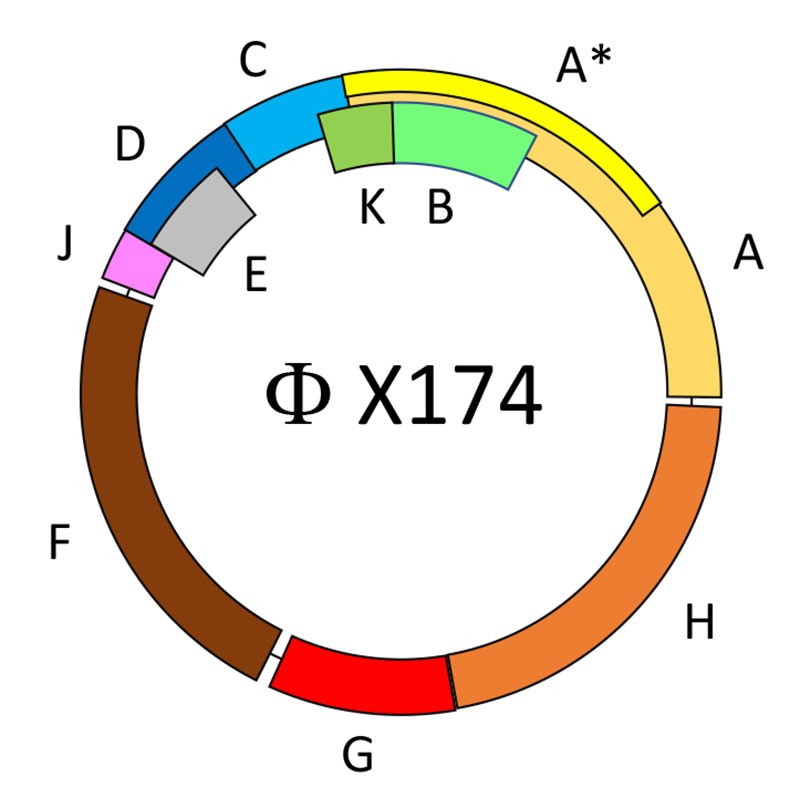
¿Cómo es posible que estos genes empalmados se originaran? Para entender por qué a los biólogos nos sorprende tanto que exista este tipo de genes, es necesario que revisemos brevemente lo que sabemos de la evolución genética.
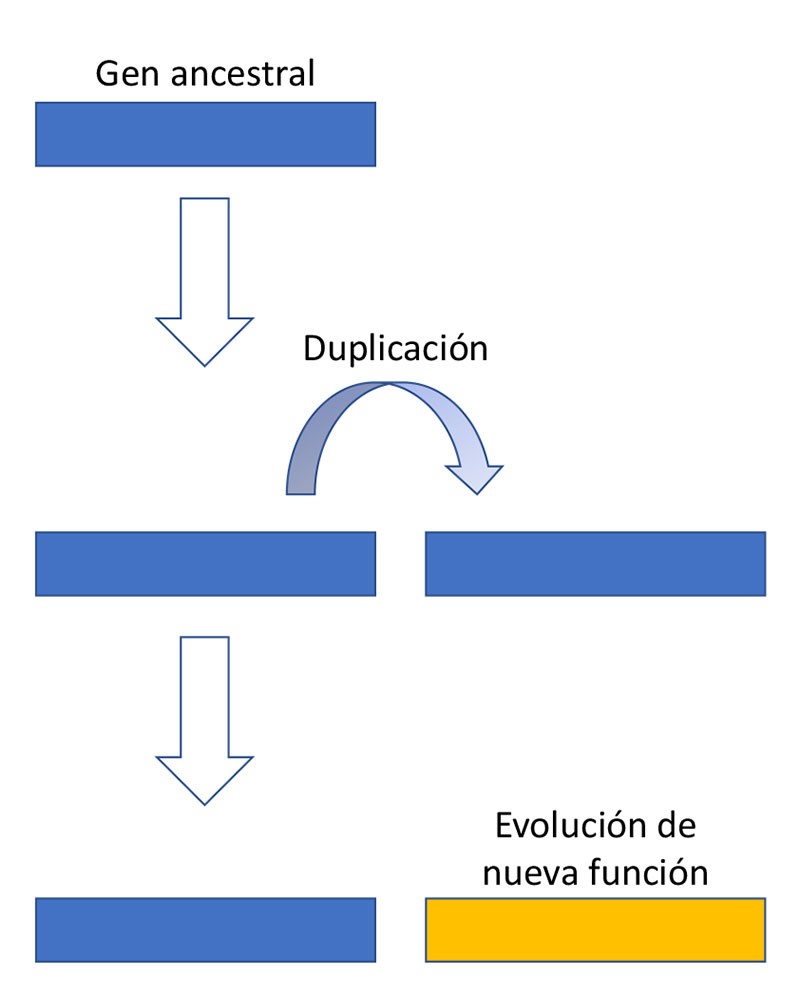
La gran mayoría de los genes se originan mediante duplicación (Figura 2). De esta forma, un gen ancestral da origen a dos genes “nuevos”. Inicialmente, las dos copias genéticas son idénticas y divergen a través de las generaciones debido a la acumulación de mutaciones. Luego, la selección natural puede favorecer el origen de una función nueva en una de las copias. En términos muy generales, este modelo describe el origen y la evolución de las familias de genes. Por ejemplo, las distintas clases de hemoglobinas que los humanos tenemos, evolucionaron de esta forma. Sin embargo, el modelo descrito no se aplica con facilidad en el caso de los genes empalmados. En 1977, el origen de ellos era un misterio sin resolver**.
Una hipótesis inverosímil
Dado que no se conocía ningún mecanismo que pudiera explicar el origen de los genes empalmados, dos investigadores decidieron poner a prueba una hipótesis sorprendente. Hiromitsu Yokoo y Tairo Oshima se preguntaron en 1979 si Phi-X174 contenía un mensaje de una civilización extraterrestre. ¡Ni más ni menos!
Estos autores hipotetizaron que una civilización extraterrestre y con una tecnología más avanzada que la nuestra sintetizó el genoma de Phi-X174 para encriptar un mensaje en uno de los genes empalmados. Supusieron que estaría codificado en la región en donde el gen A empalma con el gen B.
Las razones para elegir a este gen como recipiente del mensaje fueron dos. En primer lugar, el gen A empalma parcialmente con el gen B. Debido a ello, la tasa de evolución del gen A se ve reducida, preservando el mensaje hipotético. En segundo lugar, el gen A empalma en 121 codones con el gen B; y 121 es el resultado de multiplicar 11 x 11. ¿Por qué es relevante que 121 sea el resultado de elevar al cuadrado un número primo? Resulta que Frank Drake, un prestigioso astrónomo conocido por sus esfuerzos para buscar radioseñales de civilizaciones extraterrestres, había sugerido un sistema de comunicación interplanetario basado en matrices de números primos. Que la región empalmada entre los genes A y B fuera el resultado de multiplicar dos números primos no parecía ser una coincidencia.
La hipótesis a prueba
Con estas ideas en mente, Hiromitsu Yokoo y Tairo Oshima decidieron organizar la secuencia genética del gen A en una matriz de 11 x 11 codones (Figura 3). Basándose en la teoría de Frank Drake, los autores razonaron que el mensaje extraterrestre estaría escrito en dos dimensiones y se revelaría al transformar la matriz en una imagen.

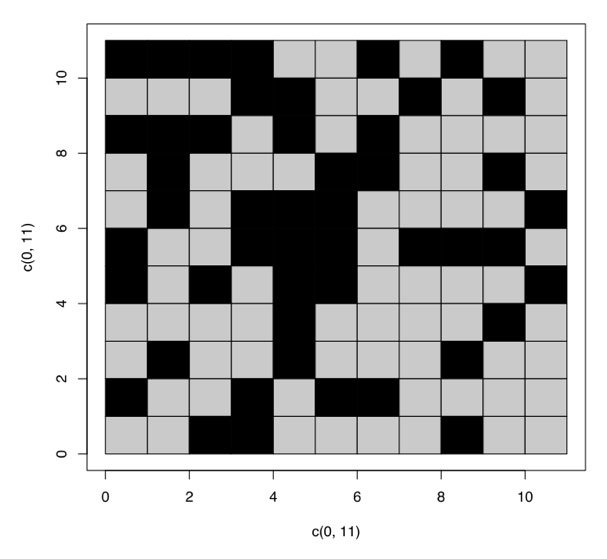
A continuación, los autores transformaron la matriz de la Figura 3 en una imagen. Para explicar cómo lo hicieron requeriremos un poco más de bioquímica. El DNA está conformado por cuatro bases: A, T, G, C. Desde el punto de vista bioquímico, las bases A y G son purinas; y las T y C son pirimidinas. Químicamente las purinas son más grandes que las pirimidinas. Hiromitsu Yokoo y Tairo Oshima tomaron una base de cada codón y transformaron las purinas en recuadros negros y las pirimidinas en blancos esperando que se formara algún patrón simple, por ejemplo, una equis. Sin embargo, al transformar la matriz en imagen sólo encontraron patrones sin sentido (Figura 4).
¿Un trabajo para el Ig Nobel?
Tal vez, a primera vista, nos podría parecer que el trabajo publicado por Hiromitsu Yokoo y Tairo Oshima podría ser merecedor de un Ig Nobel***. Recordemos que los Ig Nobel se entregan a grupos de científicos que publican trabajos que: «primero hacen reír a la gente, y luego la hacen pensar». Independientemente de ello, se trata de un artículo que cumple con todas las características de un trabajo científico. Los autores parten de una observación, plantean una hipótesis; realizan un experimento para ponerla a prueba, y, finalmente concluyen apegándose a los resultados obtenidos. Lo más importante es que esta publicación nos ofrece una oportunidad excepcionalmente clara para estudiar cómo se usan las hipótesis en la ciencia.
Clara como el agua
La primera característica de una buena hipótesis es su claridad. La de Hiromitsu Yokoo y Tairo Oshima (por descabellada que nos pueda parecer) es extremadamente clara. Los autores declaran al inicio de su artículo que:
“Se ha realizado un esfuerzo preliminar para investigar si el DNA del fago Phi-X174 posee un mensaje de una sociedad avanzada”.
Y más adelante plantean que:
“En la actualidad, es realmente difícil explicar el origen y la evolución de los genes empalmados en términos de su evolución molecular. Sin embargo, sin una explicación posible, puede ser razonable especular acerca de su origen artificial, esto es, que este DNA fue sintetizado por una tecnología muy avanzada”.
La capacidad predictiva de las hipótesis
En segundo lugar, una buena hipótesis debe de tener capacidad predictiva. Es decir, debe indicar qué deberíamos observar en caso de ser cierta. En cuanto al trabajo presentado por Hiromitsu Yokoo y Tairo Oshima, la hipótesis predice que de ser cierto que el gen A contenga un mensaje escrito por una civilización extraterrestre, deberíamos de haber encontrado un patrón no aleatorio al transformar la matriz de purinas y pirimidinas en recuadros negros y blancos.
La falsabilidad de las hipótesis
Finalmente, para que una hipótesis sea útil en la ciencia, debe de poderse falsear. Es decir, dada una hipótesis, debemos poder llevar a cabo un experimento (o una observación) que permita poner a prueba la veracidad de ésta. En el caso de Hiromitsu Yokoo y Tairo Oshima, el experimento consistió en transformar la matriz en recuadros blancos y negros esperando que se formara una imagen con algún mensaje. Al no haber encontrado ninguno, los autores concluyeron que no hallaron evidencia que soportara a su hipótesis:
“Desafortunadamente, no se observó ningún patrón significativo en los arreglos que examinamos”.
Algunas notas finales sobre las hipótesis
Los científicos construimos hipótesis para tratar de entender el mundo natural; muchas veces nos enamoramos de ellas y queremos de todo corazón que sean ciertas (en especial si somos sus autores intelectuales). Sin embargo, la forma adecuada de proceder para conocer la veracidad de una hipótesis no es tratar de probar que estas son ciertas. Por el contrario, los científicos debemos de trabajar para tratar de demostrar que nuestras hipótesis son falsas; y si no lo logramos, entonces las debemos de considerar como verdaderas temporalmente – hasta que encontremos una mejor explicación de los datos.
Por otro lado, no todos los trabajos científicos deben de tratar de poner a prueba alguna hipótesis. Muchos de ellos son exploratorios y no por ello menos relevantes. Recordemos que el método científico idealizado comienza con la observación. Por ejemplo, muchas veces, el valor de los trabajos científicos exploratorios es precisamente plantear hipótesis – que después pueden ser puestas a prueba en otros trabajos o por otros investigadores.
Pon tú a prueba la hipótesis
Si te gusta la programación, al final de este artículo viene un pequeño código en R para repetir la Figura 4. En él, puedes cambiar el valor de la variable ‘y’ para buscar un patrón utilizando otras posiciones del codón (por ejemplo, los nucleótidos 1 o 3); o bien, programar otra forma de representar las bases en la figura. Por ejemplo, en lugar de organizar las bases en purinas y pirimidinas, puedes clasificarlas en las que se aparean mediante dos (T y C) o tres (A y G) puentes de hidrógeno. Y quién sabe, tal vez seas tú quien encuentre el mensaje enviado por una civilización alienígena avanzada.
Para saber más
Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, et al. (1977) Nucleotide sequence of bacteriophage Phi-X174 DNA. Nature, 265 (5596), 687 – 695.
Yokoo, H. and Oshima, T. (1979) Is Bacteriophage ΦX174 a Message from a Extraterrestial Ingeligence? Icarus, 38: 148 – 153.
VIRUS Y LA BÚSQUEDA DE EXTRATERRESTRES, por Daniel Martínez / noviembre 7, 2016. https://ulum.es/virus-y-la-busqueda-de-extraterrestres/
Contacto (novela), https://es.wikipedia.org/wiki/Contact_(novela)
Notas
*La letra griega (Φ) o Phi, se pronuncia “fi”.
**En la actualidad conocemos el mecanismo mediante el cual los genes empalmados se originan, pero ese es tema de otro artículo de divulgación.
***Los premios Ig Nobel comenzaron a otorgarse en 1991, décadas después de la publicación de Hiromitsu Yokoo y Tairo Oshima.
Apéndice
Código en R para generar la Figura 4.
genA <- “GAATGGAACAACTCACTAAAAACCAAGCTGTCGCTACTTCCCAAGAAGCTGTTCAGAATCAGAATGAGCCGCAACTTCGGGATGAAAATGCTCACAATGACAAATCTGTCCACGGAGTGCTTAATCCAACTTACCAAGCTGGGTTACGACGCGACGCCGTTCAACCAGATATTGAAGCAGAACGCAAAAAGAGAGATGAGATTGAGGCTGGGAAAAGTTACTGTAGCCGACGTTTTGGCGGCGCAACCTGTGACGACAAATCTGCTCAAATTTATGCGCGCTTCGATAAAAATGATTGGCGTATCCAACCTGCAGAGTTTTATCGCTTCCATGACGCAGAAGTTAACACTTTCGGATATTTCT”
plot(c(0, 11), c(0,11), type = “n”)
y <- 2 # Puedes cambiar este valor a: 1, 2, 3
# para graficar distintas posiciones de cada codon
for (i in c(10:0)){
for (j in c(0:10)){
print(y)
base <- strsplit(genA, “”)[[1]][y]
print (base)
y = y +3
if (base == ‘A’){
rect( j, i, (j+1), (i+1), col=’black’)
}
if (base == ‘T’){
rect( j, i, (j+1), (i+1), col=’gray’)
}
if (base == ‘G’){
rect( j, i, (j+1), (i+1), col=’black’)
}
if (base == ‘C’){
rect( j, i, (j+1), (i+1), col=’gray’)
}
}
}