Los RNAs circulares: Todo un nuevo mundo
Los RNAs circulares (circRNAs) son una clase especial recientemente descubierta de estructuras de RNA, compuestas por secuencias derivadas de exones (secuencias que codifican para la proteína) y/o intrones (secuencias intergénicas que se eliminan durante la formación del RNA mensajero, mRNA), que son generadas por un mecanismo de retro-empalme (back splicing) y por la maquinaria del espliceosoma (fundamental para generar a los mRNAs). Durante muchos años fueron considerados como subproductos no codificantes de los mRNAs, derivados del proceso de transcripción. Estas estructuras fueron descubiertas gracias a las nuevas tecnologías de secuenciación de alto rendimiento, y a diferencia de los RNA lineales, como el mRNA (mensajero), el miRNA (micro), los snRNA (pequeños nucleares), los lncRNAs (largo no codificante) y el ncRNA (no codificante), los circRNAs no cuentan con extremos libres 3′ o 5′ debido a que son circulares. Esta característica molecular los hace más estables y resistentes a la degradación en comparación con los RNAs lineales, además de que no pueden ser reconocidos por las técnicas convencionales de secuenciación que requieren la presencia de los extremos libres 3’o 5’.
Principalmente los circRNAs se forman por el retro-empalme (back-splicing) del exón, posterior al proceso de transcripción del RNA, y tienden a acumularse en altos niveles en varios tejidos humanos, especialmente en el cerebro y están presentes en el tejido neuronal. Además, se encuentran elevados durante la neurogénesis en el humano y en los ratones, indicando que estas moléculas están involucradas en la generación de los diferentes tipos neuronales. Los circRNAs tienen un papel fundamental en la regulación transcripcional, postranscripcional y traduccional de los genes y se encuentran en diferentes especies, desde las bacterias como Escherichia coli, la mosca Drosophila melanogaster, y en otras muchas donde se les ha buscado. En el genoma humano y el de ratón se conocen más de 3,000 circRNAs, de los cuales aproximadamente 69 son ortólogos. Esta conservación evolutiva de múltiples especies, es una señal relevante de que los circRNAs podrían tener un papel esencial en un amplio espectro de funciones biológicas en los mamíferos. Los circRNAs se han detectado en diferentes tejidos humanos de los sistemas respiratorio, digestivo, endocrino, tegumentario y cardiovascular, con funciones particulares que ayudan a mantener la homeostasis de estos sistemas. Por lo tanto, la concentración alterada de los circRNAs, puede tener correlaciones con diferentes patologías humanas, como las autoinmunes, los trastornos neurológicos y el cáncer.
Formación de los circRNAs.
Los circRNAs poseen un RNA monocatenario de forma circular, catalizado por la maquinaria del espliceosoma o por las ribozimas. La unión del extremo 5′ río abajo de la molécula de RNA, con el sitio aceptor río arriba 3′, da como resultado la circularización de la molécula de RNA (Figura 1). Este mecanismo, conocido como “back-splicing” (retro-empalme), es fundamental en la generación los circRNAs. Así, pueden formarse distintos tipos de circRNAs, a través de retro-empalmes alternativos, seleccionando diferentes intrones y exones. Se ha descrito que puede existir competencia entre diversas regiones de unión al RNA, lo que podría conducir a la formación de circRNAs con exones distintos, a partir de un solo pre-mRNA (el RNA que contiene exones e intrones).
Dependiendo de su origen, el circRNA puede componerse de exones, intrones o estructuras exón-intrón (ElciRNA). El circRNA exónico (ceRNA) se acumula principalmente en el citoplasma y es resultado del proceso de empalme de las regiones exónicas del pre-mRNA. (Fig. 1). Este desarrollo puede abarcar uno o más exones que dependen de la región de circularización del pre-mRNA. La dimensión del exón definirá la estructura final del circRNA exónico, siendo el tamaño promedio de aproximadamente 404 a 709 nucleótidos (nt). El circRNA intrónico (ciRNA) se produce a partir de las estructuras “lariats” derivadas del splicing, resistentes a la degradación por exonucleasas (Fig. 1).
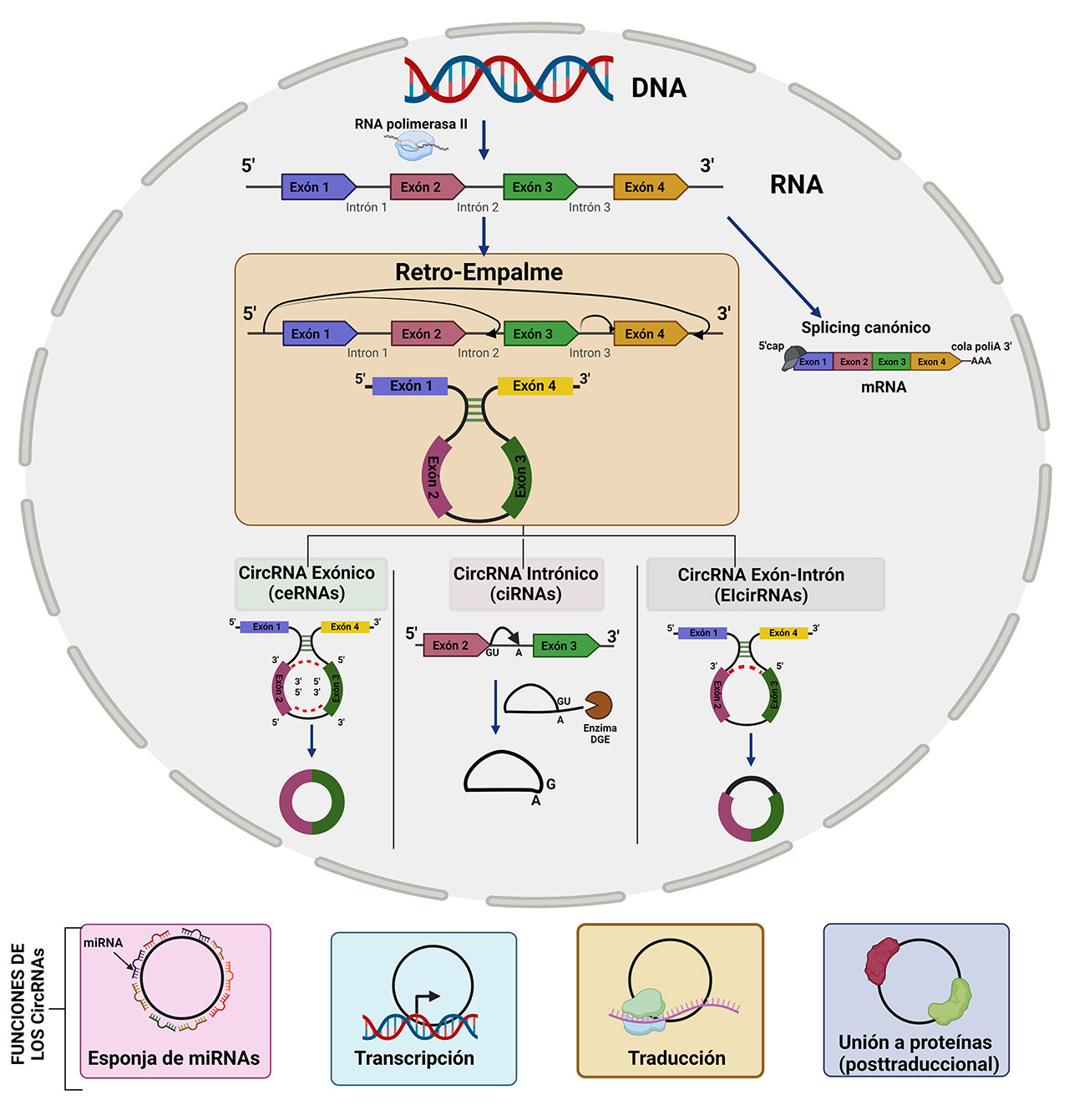
Una de las diferencias moleculares más relevantes del circRNA intrónico es la presencia de un enlace de carbono 2′-5′ en el punto de ramificación de empalme, mientras que el exónico se compone principalmente de una estructura fosfodiéster de 3′-5′. Otra forma de circRNAs es una combinación de exones e intrones dentro de la misma molécula circularizada. Aunque inusual, algunos fragmentos de intrones también pueden estar en la conformación del circRNA. Esta estructura se denomina circRNA exón-intrón (o ElciRNAs) (Fig. 1). Este tipo de circRNA se forma por un mecanismo similar al de los circRNAs exónicos, pero sin el corte y la presencia de sitios de empalme canónicos para la eliminación de los intrones. En el genoma humano los genes productores de circRNAs tienden a ser más largos y contienen más exones que los genes promedio. Una de las funciones establecidas para los circRNAs, es la capacidad de actuar como esponjas de los miRNAs, que son moléculas pequeñas de RNA capaces de regular al 30% de los genes que codifican proteínas, y por lo tanto, influir en las acciones transcripcionales que tienen estas moléculas. Para realizar esta función, los circRNAs presentan múltiples sitios de unión para los miRNAs, lo que les permite capturarlos y controlar indirectamente la expresión génica.
Las proteínas de unión al RNA (RBP, por sus siglas en inglés), también pueden servir como factores reguladores en la formación de los circRNAs. Un ejemplo es la RBP proteína temblorosa (QKI), que regula la formación de circRNA mediante la unión a su pre-mRNA, que presenta sitios de unión capaces de inducir el retro-empalme y, en consecuencia, la circularización de la molécula. Otro regulador importante en la síntesis de los circRNAs es la proteína adenosina desaminasa 1 (ADAR-1). Un incremento en esta enzima regula a la baja la expresión de los circRNAs. El contexto genético juega un papel esencial en la expresión y biogénesis del circRNA, mostrando una expresión específica de tejido.
El propósito real de esta molécula en la homeostasis humana sigue siendo desconocido. Aunque se sabe que pueden actuar como esponjas, su correlación con diferentes tejidos podría ayudarnos a entender mejor sus funciones. A pesar de que los circRNAs están presentes en numerosos tejidos del humano, el tejido neuronal tiene una abundante expresión de estas moléculas.
En el cerebro de los mamíferos, los circRNAs se elevan en diferentes regiones, especialmente en el cerebelo, y muestran una expresión diferente que depende de la etapa del desarrollo. Los cambios epigenéticos (variación en la actividad y expresión génica, sin cambiar las secuencias de DNA), también influyen en la biogénesis de los circRNA. Estas transcripciones circulares tienden a acumularse durante el envejecimiento en varios tejidos, especialmente en el cerebro y su expresión se correlaciona con la aparición de varias patologías, incluido un amplio espectro de trastornos neurológicos. Los hallazgos anteriores también han demostrado que los circRNAs tienen una presencia significativa en el tejido neuronal y se encuentran elevados durante la neurogénesis, con un número importante derivado de genes neuronales, lo que sugiere que estas moléculas circulares están implicadas en el fenotipo celular y molecular de nuestro cerebro. Sin embargo, la biogénesis completa de los muchos tipos de moléculas circRNAs no está completa aún y su participación en el fenotipo neuronal y el desarrollo de patologías siguen siendo un tema desafiante para los científicos.
Aunque existe una distribución asimétrica de los circRNAs en las neuronas, se ha observado que se encuentran en mayor concentración en las terminales sinápticas, actuando como una molécula con funcionalidad específica en las sinapsis. Al ser más estables que los RNA lineales, los circRNAs podrían actuar en estados de estrés y también funcionar como compuestos relacionados con la memoria. Además, durante la diferenciación neuronal, el número de circRNAs aumenta significativamente. Por lo tanto, muchos circRNAs se expresan en diferentes momentos de diferenciación y maduración neuronal, especialmente en el desarrollo postnatal. La expresión ectópica de ciRS-7 deriva en un desarrollo anormal del mesencéfalo en el pez cebra. En nuestro cerebro, el circRNA más abundante es ciRS-7 y contiene más de 70 sitios de unión para el miRNA-7 (miR-7), reprimiendo su funcionamiento normal, lo que supone un papel esencial en el desarrollo del cerebro. La investigación actual muestra que los circRNAs se enriquecen en las neuronas y se acumulan en la corteza cerebral y el hipocampo en los ancianos, lo que sugiere que su sobrecarga puede conducir a la disfunción celular y cerebral. Por lo tanto, a medida que el individuo avanza en edad, los niveles de expresión de los circRNAs aumentan en el sistema nervioso y pueden conducir al desarrollo de varias enfermedades neurológicas.
Por ejemplo, en el tejido cerebral de pacientes con Enfermedad de Alzheimer (EA), se ha demostrado que se encuentra disminuido ciRS-7, ocasionando un aumento de miRNA-7 en el neocórtex y el hipocampo. Este aumento en miRNA-7 provoca una disminución de la enzima conjugadora de ubiquitina E2A (UBE2A), que es una proteína fagocítica autofágica esencial para que el cerebro pueda eliminar los péptidos amiloides y otras moléculas relacionadas con efectos citotóxicos producidos por la degeneración progresiva del sistema nervioso central.
Por otro lado, ciRS-7 también se asocia con la Enfermedad de Parkinson (EP) en la cual hay un desequilibrio entre las tasas de síntesis, agregación y eliminación de alfa-sinucleína (α-syn) resultando en la formación de especies oligoméricas y fibrilares tóxicas. Sin embargo, los mecanismos moleculares a través de los cuales, los agregados anormales de α-syn contribuyen a la neurodegeneración en la EP no son claros. Se sabe que miR-7 regula negativamente la expresión de α-syn, al unirse directamente al extremo 3′-UTR de su mRNA. Clínicamente, los pacientes con EP tienen niveles significativamente más bajos de miR-7 en la sustancia nigra, y como se mencionó, ciRS-7 puede actuar como esponja de miR-7 y atraparlo. De esta manera, la sobreexpresión de ciRS-7 podría reducir los niveles de miR-7, lo que lleva a un aumento en la expresión cerebral α-syn.
En los trastornos del espectro autista (TEA), caracterizados por el deterioro de la interacción social, la comunicación verbal y no verbal y el comportamiento repetitivo, las investigaciones recientes han encontrado alrededor de 60 circRNAs, expresados diferencialmente en los cerebros post-mortem de individuos con TEA. Este estudio también investigó la correlación de los circRNAs con la expresión de miRNAs y mRNA en muestras de corteza de pacientes con TEA, y halló 8,170 interacciones circRNA-miRNA-mRNA. Estos resultados sugieren que varios genes de riesgo de TEA, como la neuroligina 1 (NLGN1), el antígeno estromal 1 (STAG1), el hidroxiesteroide 11-beta deshidrogenasa 1 (HSD11B1), el péptido intestinal vasoactivo (VIP) y la enzima activadora 6 modificadora similar a la ubiquitina (UBA6), están regulados por un mecanismo de circRNAs.
En un modelo de daño cerebral por isquemia-hipoxia (HIBD) para ratas, sometidas a isquemia, ligando permanentemente la arteria carótida derecha. Después de un tiempo de recuperación de 1 h, los animales son expuestos a hipoxia en una cámara con 8% O2 y 92% de N2 durante 2.5 h y luego regresan a su vida normal. En el cerebro, se identificaron alrededor de 66 circRNAs expresados diferencialmente (20 regulados al alza y 46 a la baja) en comparación con el grupo control. Además, estos circRNA alterados, fueron 70% de origen exónico y un 28% de origen intergénico. Estos resultados suponen que la isquemia causada por la falta de oxígeno en el cerebro puede estar directamente vinculada con el desequilibrio de la expresión de los circRNAs en la corteza. Otro RNA circular con característica de esponja implicado en los tejidos neuronales es el circRNA Sry. Esta molécula tiene sitios de unión que pueden capturar miR-138 y se asocia con procesos cognitivos como el aprendizaje y la memoria. En otro estudio se evaluó el efecto del circRNA en el trastorno bipolar, con alteraciones significativas de la expresión de dos circRNA específicos, cNEBL y cEPHA3, presentes en los loci genómicos de los genes NEBL (Nebulette) y EPH Receptor A3 (EPHA3).
Desde luego, la función de los circRNAs requiere más investigaciones, y aunque el campo de la transcriptómica ha permitido un desarrollo creciente en la detección de los circRNAs, es necesario profundizar en su identificación y clasificación, principalmente para detectar posibles variaciones entre estas moléculas. La asociación de circRNAs con varios fenotipos neurológicos y con distintas enfermedades ya han demostrado la importancia que estas moléculas pueden tener como elementos centrales de la patogénesis en el tejido neurológico. Sin embargo, el estudio del circRNA para patologías humanas aún tiene mucho por avanzar, pero puede ayudar a dilucidar dudas y guiar a los científicos en este nuevo y apasionante campo del RNA.
Referencias:
- Guerra BS, et al. Biogenesis of circular RNAs and their role in cellular and molecular phenotypes of neurological disorders. Semin Cell Dev Biol. 2021 Jun;114:1-10. doi: 10.1016/j.semcdb.2020.08.003. Epub 2020 Sep 3. PMID: 32893132.
- Han B, Chao J, Yao H. Circular RNA and its mechanisms in disease: From the bench to the clinic. Pharmacol Ther. 2018 Jul;187:31-44. doi: 10.1016/j.pharmthera.2018.01.010. Epub 2018 Feb 14. PMID: 29406246.
- Hanan M, Soreq H, Kadener S. CircRNAs in the brain. RNA Biol. 2017 Aug 3;14(8):1028-1034. doi: 10.1080/15476286.2016.1255398. Epub 2016 Nov 28. PMID: 27892769; PMCID: PMC5680707.