Sistema de transporte nuclear de las importinas alfa y beta en Entamoeba histolytica
Introducción
Entamoeba histolytica es un parásito protozoario perteneciente al filo amoebozoa y es conocido por ser el agente etiológico de la disentería amebiana, conocida como amebiasis en humanos. De manera silvestre E. histolytica experimenta un ciclo de vida complejo que involucra a un hospedero (Figura 1). El 90% de los pacientes infectados presentan síntomas y pueden mantener los parásitos en el lumen del colon, mientras que el 10% restante no muestra síntomas (Begum et al., 2021). En el mundo, aproximadamente 50 millones de personas son infectadas anualmente, con más de 100,000 muertes debido a la disentería amebiana (Zulfiqar et al., 2023). En 2022 en México se reportaron más de 162,000 casos de amebiasis intestinal y 428 casos de abscesos hepáticos causados por la infección con este parásito protozoario (Revista Epidemiológica Nacional).
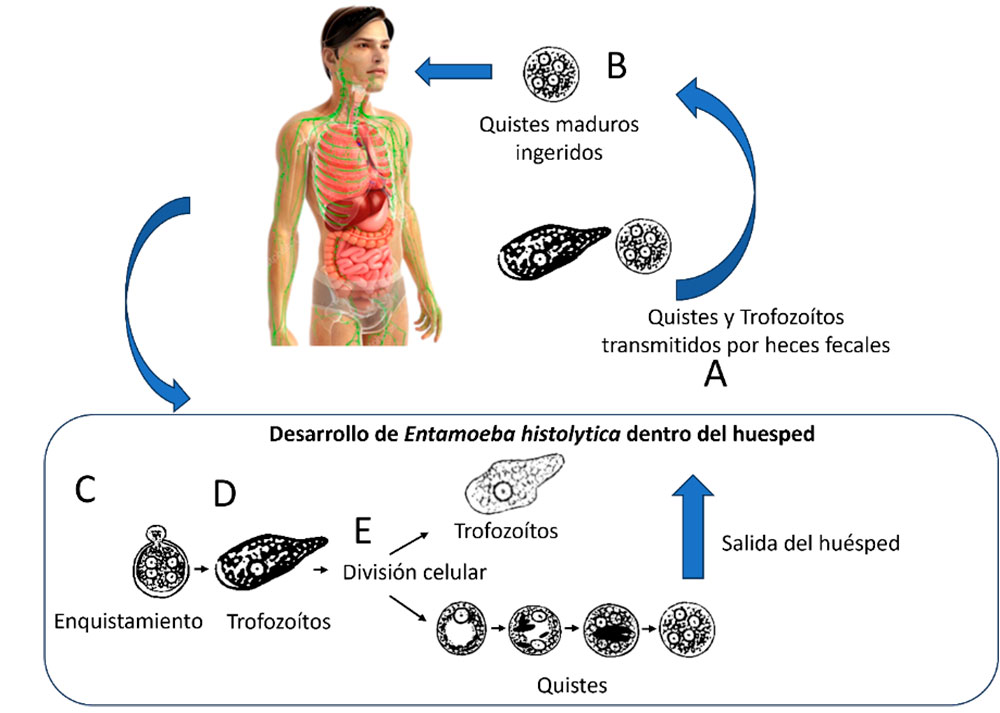
Ciclo de vida de E. histolytica
Los quistes y los trofozoítos son transmitidos principalmente por la materia fecal. Los quistes suelen estar presentes en las heces sólidas mientras que los trofozoítos están en el material diarreico. La infección con E. histolytica ocurre principalmente mediante la ingestión de alimentos o agua contaminada con quistes maduros (Marie and Petri, 2013; Ralston, 2015). También se han reportado infecciones mediante contacto sexual oro-anal. Una vez dentro del huésped, el enquistamiento se lleva a cabo en el intestino delgado y los trofozoítos son liberados para posteriormente migrar al intestino grueso. Se puede presentar una infección no invasiva en la cual los trofozoítos permanecen en el lumen intestinal sin que el huésped presente algún tipo de sintomatología. Los trofozoítos se dividen por fisión binaria y son capaces de infectar la mucosa intestinal, los vasos sanguíneos y sitios extraintestinales como cerebro, pulmones e hígado. Los quistes están adaptados para sobrevivir de días a semanas y permanecer en un estado infeccioso debido a que sus paredes celulares le confieren resistencia al medio externo. De manera contrastante, los trofozoítos no resisten al medio exterior una vez que son expulsados del huésped, y si son ingeridos oralmente, pueden no resistir a los ácidos gástricos producidos en la digestión estomacal. Los síntomas clínicos incluyen dolor abdominal, pérdida de peso, diarrea o inclusive diarrea con sangre, que puede durar varias semanas. Una infección sistémica puede perdurar e incluso años después de la infección inicial, en la que el órgano comúnmente más afectado es el hígado en el que se forman abscesos hepáticos; también se puede presentar ictericia, fiebre, tos, escalofríos, aumento en el volumen del hígado y pérdida de peso (Haque et al., 2003; Pritt and Clark, 2008).
Patogenicidad
Los mecanismos de patogenicidad de E. histolytica incluyen adherencia, citotoxicidad y fagocitosis/trogocitosis. La adherencia es un proceso fundamental para la infección de E. histolytica. La secreción de cistein-proteasas ocasiona el adelgazamiento de las mucinas del colon del huésped y permite a los trofozoítos unirse a las capas de mucina por medio de la vía de Gal/lectina. Posteriormente, los trofozoítos son capaces de unirse a las células epiteliales del huésped (Begum et al., 2015; Singh et al., 2016).
Después de la adherencia, se puede entrar a un proceso en el que E. histolytica mata a las células del huésped y a las bacterias intestinales, haciendo interacción directa entre el parásito con las células del huésped y la secreción de proteinasas para facilitarle la infección que puede desatar un proceso de activación de apoptosis (Christy and Petri, 2011; Becker et al., 2010). Este parásito protozoario codifica para proteínas llamadas amebaporos que pueden formar poros en las bicapas lipídicas cuya función es la fagocitosis de material extracelular por medio de fagosomas (Berninghausen y Leippe, 1997; Ralston, 2015).
En años recientes se ha fortalecido la hipótesis de que la trogocitosis es el principal mecanismo que utiliza E. histolytica para engullir y alimentarse de células vivas (Ralston et al., 2014). Para unirse a las células vivas del huésped E. histolytica lleva a cabo un rearreglo de los filamentos de actina y posteriormente va engullendo pequeñas porciones de las células del huésped, ocasionando una muerte celular que conduce a una disociación en la interacción de E. histolytica con la célula huésped (Ralston, 2014; Ralston, 2015).
El proceso de fagocitosis es crucial para la obtención de nutrientes para E. histolytica al invadir los tejidos del huésped y al interactuar las partículas con los receptores de superficie celular del parásito. Este fenómeno se lleva a cabo debido a un rearreglo de los microfilamentos de actina que proveen la formación del fagosoma para el engullimiento del material extracelular (Christy y Petri, 2011). La actina es una proteína fundamental en los fagosomas tempranos, ya que están rodeados de filamentos de actina que se van despolimerizando gradualmente conforme el fagosoma va madurando. Esta maduración se ve controlada por proteínas pertenecientes a la superfamilia Ras, principalmente las proteínas Rab, fosfatidilinositol y proteínas cinasas (Saito-Nakano et al., 2005; Somlata et al., 2011; Somlata et al., 2012; Avalos-Padilla et al., 2015; Verma et al., 2015; Verma et al., 2016; Hanadate et al., 2016). Similar a eucariontes multicelulares, parece que este proceso se rige por el complejo ESCRT (del inglés: Endosomal Sroting Complexes Required for Transport) (Lopez-Reyes, 2010). Adicionalmente se ha visto que el Ca2+ está implicado en el proceso de fagocitosis, principalmente en la formación de fagosomas en donde han sido identificadas algunas proteínas incluidas en este proceso como son: EhCaBP3 y EhCaBP1 que se localizan de manera ubicua en el cuerpo del parásito (Sahoo et al., 2004).
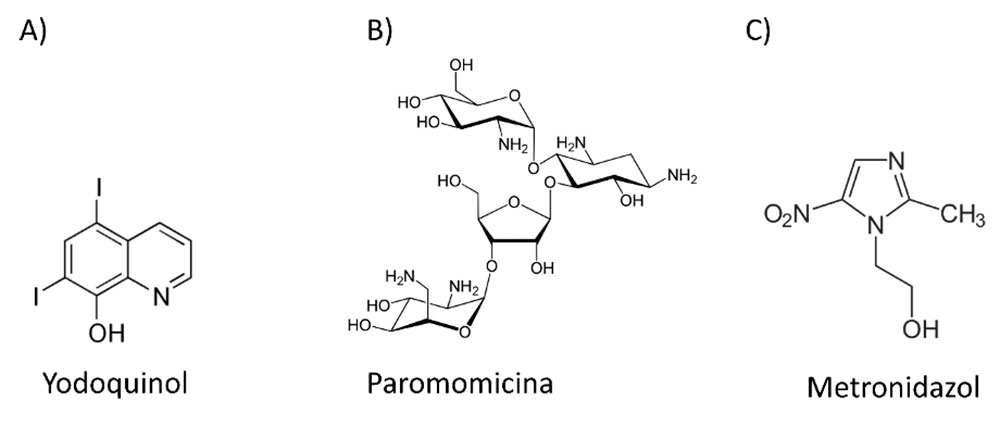
Tratamientos actuales
La invasión intestinal asintomática con E. histolytica, frecuentemente se trata con yodoquinol y paromomicina, mientras que el tejido invadido es tratado con metronidazol (Figura 2); sin embargo, el metronidazol es un antibiótico no especifico usado para combatir bacterias anaerobias e infecciones por protozoarios. Además, el metronidazol tiene efectos cardiovasculares adversos y problemas gastrointestinales (Kimura et al., 2007; Stanley, 2003). Existen pocos estudios con drogas distintas al metronidazol, extractos herbales en pacientes con amebiasis o con una droga llamada auranofin cuyo uso es para tratar la artritis reumatoide. Se sabe que auranofin es una droga cuyo blanco es la tiorredoxina reductasa que previene la reducción de tiorredoxina y potencia la sensibilidad de los trofozoítos al reaccionar con el oxígeno (Sha et al., 2016, Debnath et al., 2012). Actualmente se desarrollan moléculas que inhiben el transporte nuclear para el tratamiento de infecciones virales como el VIH y el Dengue, y se ha probado el uso de ivermectina para inhibir el trasporte nuclear de la RNA Pol I en Trypanosoma cruzi los cuales se podrían probar para la disentería amebiana; sin embargo, se requiere de la investigación farmacológica y de biología molecular en E. histolytica (Wagstaff et al., 2012; Canela-Pérez et al., 2018). En particular, se necesita del conocimiento de los sistemas de transporte de proteínas hacia y desde el núcleo para evaluar la factibilidad de uso de fármacos como la ivermectina para tratar la amibiasis.
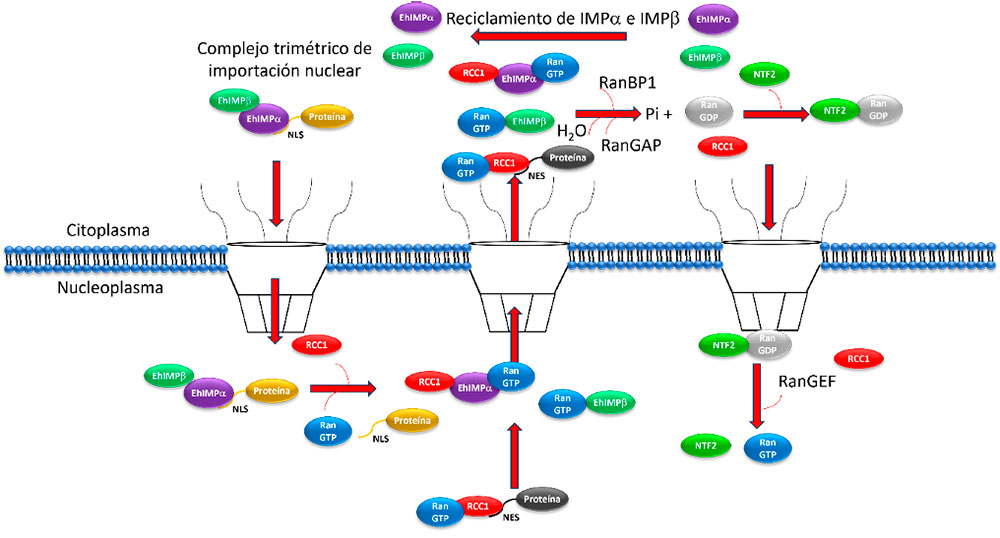
Transporte nuclear de proteínas mediado por Importina α e Importina β
Es bien sabido que la vía de importación nuclear α/β está bien conservada en eucariontes, como Saccharomyces cerevisiae, Mus musculus y Homo sapiens (Wente and Rout et al., 2010). Existe poca literatura de esta vía de transporte nuclear de proteínas en protozoarios, por lo tanto, la vía de transporte nuclear canónica α/β que involucra a la importina alfa (IMPα) y beta (IMPβ), no ha sido estudiada en E. histolytica. Sin embargo, hemos realizado una búsqueda informática y encontramos anotados los genes codificantes para estas proteínas en la base de datos AmoebaDB (números de acceso: EHI_025350 para IMPα y EHI_036520 para IMPβ).
El transporte nuclear de proteínas es un proceso altamente complejo que transcurre a través de los complejos de poro nuclear (NPC). En esta vía participan proteínas llamadas importinas y exportinas pertenecientes a la familia de las carioferinas (Strambio-De-Castillia et al., 2010). El mecanismo de translocación de proteínas al nucleoplasma mediado por IMPα e IMPβ se conoce como vía clásica de importación nuclear de proteínas (Cauntain et al., 2015). Las proteínas nucleares son transportadas desde el citoplasma al interior de núcleo (Cargo) y generalmente tienen motivos proteínicos conocidos como señales de localización nuclear (NLS), que son reconocidos por la proteína adaptadora IMPα formando el complejo Cargo/IMPα; posteriormente la proteína receptora IMPβ se une al dominio IBB (Importin Beta Binding domain) de IMPα formando el complejo trimérico de transporte Cargo/IMPα/IMPβ (Stewart y Rhodes, 1999; Stewart, 2007). Este complejo es translocado al nucleoplasma atravesando el NPC mediado por IMPβ al interactuar con dominios ricos en fenilalanina y glicina (FG) de las nucleoporinas que conforman el NPC (Ma et al., 2016). Una vez que el complejo trimérico se encuentra en el nucleoplasma, una proteína de unión a GTP llamada Ran-GTP (perteneciente a la familia RAS) se une a IMPβ generando un cambio conformacional y así disgregando el complejo trimérico Cargo/IMPα/IMPβ (Vetter et al., 1999). IMPβ permanece unida a Ran-GTP y es exportada al citoplasma; así, Ran-GTP cumple una función regulatoria del transporte nuclear al mantener un ciclo de unión con GTP y GDP, siendo el gradiente de Ran-GTP mayoritario en el núcleo (Görlich et al., 1996; Hoelz y Blobel, 2004). La unión de GTP a Ran es modulada por el factor de intercambio de nucleótido de guanina (RanGEF) en el núcleo y la proteína activadora de GTPasa de Ran (Ran GAP), ambas proteínas distribuidas de manera asimétrica entre el núcleo y citoplasma controlan la unión de Ran a GTP o GDP y por consiguiente, la regulación del transporte nuclear (Bischoff et al., 1994; Moroianu et al., 1996; King et al.,2006). Por otra parte, IMPα es transportada al citoplasma mediante la proteina CAS (del inglés: the Celular Apoptosis Susceptibility gene product), en complejo con RanGTP (Kutay et al., 1997; Stewart, 2007). Los complejos exportados RanGTP-CAS-IMPa y RanGTP-IMPβ son disociados por la hidrólisis de RanGTP generando RanGDP en el citoplasma. La actividad de GTPasa de Ran es estimulada por la proteína activadora de GTPasa RanGAP1 (Seewald et al., 2002). Adicionalmente, las proteínas RanBP1 o RanBP2 estimulan a RanGAP1 para la activación de GTPasa de Ran (Matunis et al., 1996; Vetter et al., 1999; Ritterhoff et al., 2016). El transporte nuclear de RanGDP depende de NTF2 (Nuclear Transport Factor 2) que tiene afinidad por los dominios FG de las nucleoporinas que conforman el NPC (Stewart, 2000; Jovanovic-Talisman et al., 2009). Una vez en el nucleoplasma NTF2-RanGDP es disociado por la reconversión de RanGDP a Ran-GTP realizado por RanGEF (Bischoff y Ponstingl, 1991). Mientras que la proteína CAS puede regresar nuevamente al núcleo por sí misma. Las proteínas exportadas al citoplasma contienen un motivo llamado señal de exportación nuclear (NES) que es reconocido por factores de exportación nuclear llamados exportinas; estas son proteínas pertenecientes también a la familia de las carioferinas. El proceso de exportación nuclear requiere la participación de Ran-GTP que se une a las exportinas (Xu y Massagué, 2004) (Figura 3).
En E. histolytica, la vía de transporte nuclear clásica no ha sido estudiada; sin embargo, en la base de datos AmoebaDB identificamos los genes que codifican para las proteínas lMPα, IMPβ, CAS y Ran, RanGEF, RanBP1 y RanGAP, indicando que la vía se encuentra conservada en este parásito protozoario, similar a lo que ocurre en tripanosomatidos (Canela-Pérez et al., 2019). Se ha reportado que en los genomas de L. major y T. brucei se han identificado las proteínas NTF2, RanGAP, RanBP1 y RCC1 (Casanova et al., 2008).
Importina α
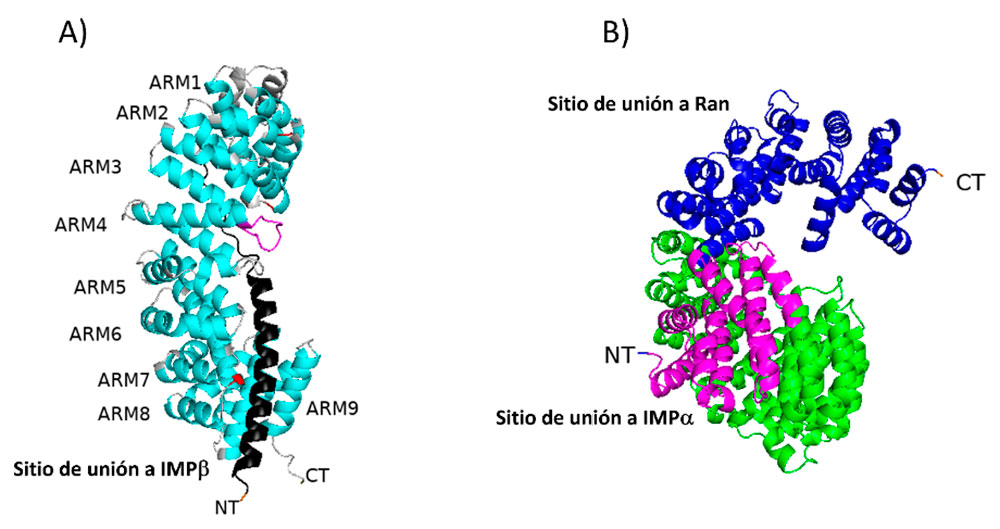
IMPα es una proteína perteneciente a la familia de las carioferinas que participa como adaptador en la vía clásica de importación nuclear de proteínas, al reconocer las cNLS de las proteínas cargo (Goldfarb et al., 2004; Oka y Yoneda, 2018). En su extremo amino muestra el dominio IBB y en su extremo carboxilo presenta 10 dominios Armadillo (ARM) (Cingolani et al., 1999; Kobe, 1999). Los dominios ARM fueron descritos en D. melanogaster en la proteína Armadillo y se conforman por tres hélices a (H1, H2 y H3) (Riggleman et al.,1989). Los dominios ARM generan una estructura conocida como alfa solenoide constituida por motivos hélice-giro-hélice (Fournier et al., 2013) que giran consecutivamente en la estructura de IMPα creando un surco a lo largo del eje de la superhélice de la proteína en donde se localizan los sitios de unión a las cNLS, específicamente los motivos conservados (^R/K) XXWXXXN), en donde ^R/K es cualquier residuo a excepción de Arg o Lys (principalmente residuos ácidos o hidrofóbicos), mientras que X es cualquier aminoácido (Conti et al., 1998; Conti y Kuriyan, 2000). Los residuos encargados de la interacción con la cNLS son Asn y Trp que generan interacción con Arg y Lys (Fontes et al., 2000). Las cNLS monopartitas y bipartitas se unen a los sitios de unión de IMPα en una conformación extendida (Fontes et al., 2003). Mediante análisis estructurales se ha descrito que las cNLSs monopartitas se unen al bolsillo mayor de IMPα, mientras que las cNLS bipartitas se unen a los bolsillos menor y mayor (Yang et al., 2010). Por otra parte, las mutaciones en residuos en ambos bolsillos de manera simultanea interrumpen la unión de NLS bipartitas (Leung et al., 2003). A la fecha, se ha logrado determinar la estructura de IMPα de H. sapiens (Dias et al., 2009), S. cerevisiae (Conti y Kuriyan, 2000), M. musculus (Kobe, 1999; Fontes et al., 2000), Oryza sativa (Chang et al., 2012; Chang et al., 2014), Arabidopsis thaliana (Wirthmueller et al., 2015) y Neurospora crassa (Bernardes et al., 2015).
IMPα no ha sido estudiada en E. histolytica, sin embargo, la hemos modelado usando el programa alphafold en alphafold.ebi.ac.uk y buscamos sus dominios con el software Pfam que se resaltan en la Figura 4A. IMPα también se ha estudiado en tripanosomátidos y se encuentra anotada en la base de datos TriTrypDB con los números de acceso de T. cruzi (TcCLB.509965.110), T. brucei (Tb927.6.2640) y L. major (LmjF.30.1120) (Canela-Pérez et al., 2020).
Importina β
Importina beta es una proteína receptora soluble que se une a Imporina alfa unida a la proteína cargo con una NLS. Esta proteína pertenece a la familia de las carioferinas y se une mediante su extremo amino al extremo amino de importina alfa. La IMPβ de E. histolytica no ha sido estudiada, sin embargo, se ha podido dilucidar la estructura completa en modelo murino (Cingolani et al., 1999). IMPβ está constituida por dominios HEAT consecutivos que conforman una super hélice. Hacia su extremo carboxilo tiene el sitio de unión a Ran GTP, que al unirse genera un cambio alostérico en IMPβ para liberar al complejo trimérico de importación IMPα/IMPβ/Cargo en el nucleoplasma. La plasticidad estructural de IMPβ y su sustrato IMPα es una característica general de las carioferinas (Chook and Blobel; 2001). Se ha descrito que algunas proteínas pueden unirse directamente a IMPβ sin la necesidad de IMPα, como es el caso de la proteína relacionada a la hormona paratiroide (PTHrP) uniéndose a su región amino de la misma manera que la ciclina B1 (Lam et al., 1999; Moore et al., 1999). Estos datos sugieren que IMPβ puede tener más de un sustrato y por lo tanto reconocer diversos tipos de NLSs. Asimismo logramos modelar IMPβ de E. histolytica usando el programa alphafold en la que se resaltan los dominios de unión a IMPα y Ran-GTP (Figura 4B).
La vía de importación nuclear de proteínas de las importinas α/β es un terreno inexplorado en E. histolytica.
Hasta la fecha no se ha estudiado la via α/β de importación de proteínas nucleares en E. histolytica, por lo que nuestra búsqueda de los genes codificantes para IMPα e IMPβ en la base de datos AmoebaDB, abre las puertas a explorar esta vía hasta ahora sin estudiar en este parásito protozoario. Nuestros modelos estructurales indican una alta conservación estructural con las proteínas ortólogas de S. cerevisiae y tripanosomátidos, en donde observamos que se mantienen presentes los dominios principales. Los números de acceso para EhIMPα es EHI_025350 y para EhIMPβ EHI_036520, respectivamente. Nuestros datos indican que esta vía de importación nuclear se encuentra conservada en E. histolytica y en otros parásitos protozoarios como Trypanosoma cruzi y Trypanosoma brucei en donde se han logrado estudiar e identificar estos componentes (Canela-Pérez et al., 2019; Canela-Pérez et al., 2020). La conservación de los factores de esta vía de importación nuclear en eucariontes unicelulares, nos indica un surgimiento temprano en el proceso de importación nuclear de proteínas en una etapa inicial de la evolución de los eucariontes.
Conclusión
La vía de transporte nuclear clásica en E. histolytica es un campo inexplorado, por lo que la identificación de las importinas α y β resultan una contribución muy importante al campo del conocimiento científico en este protozoario. La estructura molecular de ambas importinas nos indica una conservación estructural al ser comparada con otros organismos como levaduras, trypanosomas y plantas, resultando necesaria su cristalización para poder ahondar en estudios de comparación evolutiva y filogenia. Será crucial la investigación de fármacos que puedan bloquear de manera específica el mecanismo de importación y exportación nuclear de proteínas en E. histolytica.
Referencias
- Conti E, Uy M, Leighton L, Blobel G, Kuriyan J. (1998), Crystallographic analysis of the recognition of a nuclear localization signal by the nuclear import factor karyopherin alpha. Cell. 94(2):193-204. DOI:10.1016/s0092-8674(00)81419-1.
- Fontes MR, Teh T, Kobe B. (2000), Structural basis of recognition of monopartite and bipartite nuclear localization sequences by mammalian importin-alpha. J Mol Biol.297(5):1183-94. DOI:10.1006/jmbi.2000.3642.
- Avalos-Padilla, Betanzos A., Javier-Reyna R., García-Rivera G., Chávez-Munguía B., Lagunes-Guillén A., Ortega J., Orozco E. (2015). EhVps32 Is a Vacuole-Associated Protein Involved in Pinocytosis and Phagocytosis of Entamoeaba histolytica. PLoS Pathog. 11(7):e1005079. doi: 10.1371/journal.ppat.1005079.
- Becker SM, Cho KN,* Xiaoti Guo X,* Kirsten Fendig K,† Oosman MN, Whitehead R, Cohn SM, Houpt (2010). Epithelial Cell Apoptosis Facilitates Entamoeba histolytica Infection in the Gut. Am J Pathol. 176(3): 1316–1322. doi: 10.2353/ajpath.2010.090740
- Begum S, Gorman H, Chadha A, Chadee K. (2021). Entamoeba histolytica. Trends Parasitol. 37(7):676-677. doi: 10.1016/j.pt.2021.01.001.
- Begum, Quach J., Chadee K. (2015). Immune Evasion Mechanisms of Entamoeba histolytica: Progression to Disease. Front Microbiol. 6: 1394. doi: 10.3389/fmicb.2015.01394.
- Bernardes NE, Takeda AA, Dreyer TR, Freitas FZ, Bertolini MC, Fontes MR. (2015). Structure of Importin-α from a Filamentous Fungus in Complex with a Classical Nuclear Localization Signal. PLoS One.10(6):e0128687. DOI:10.1371/journal.pone.0128687.
- Berninghausen, Leippe M., (1997). Necrosis versus apoptosis as the mechanism of target cell death induced by Entamoeba histolytica. Infect Immun. 65(9):3615-21.doi: 10.1128/iai.65.9.3615-3621.1997.
- Bischoff FR, Klebe C, Kretschmer J, Wittinghofer A, Ponstingl H. (1994), RanGAP1 induces GTPase activity of nuclear Ras-related Ran. Proc Natl Acad Sci U S A. 91(7):2587-91. DOI:10.1073/pnas.91.7.2587.
- Bischoff FR, Ponstingl H. (1991), Catalysis of guanine nucleotide exchange on Ran by the mitotic regulator RCC1. Nature. 354(6348):80-2. DOI:10.1038/354080a0.
- Canela-Pérez I, López-Villaseñor I, Cevallos AM, Hernández R. (2018). Nuclear distribution of the Trypanosoma cruzi RNA Pol I subunit RPA31 during growth and metacyclogenesis, and characterization of its nuclear localization signal. Parasitol Res.117(3):911-918. DOI:10.1007/s00436-018-5747-4.
- Canela-Perez, I.; Lopez-Villasenor, I.; Cevallos, A.M.; Hernandez, R. Trypanosoma cruzi Importin alpha: ability to bind to a functional classical nuclear localization signal of the bipartite type. Parasitol Res 2020, 119, 3899-3907, doi:10.1007/s00436-020-06885-z.
- Canela-Perez, I.; Lopez-Villasenor, I.; Mendoza, L.; Cevallos, A.M.; Hernandez, R. Nuclear localization signals in trypanosomal proteins. Mol Biochem Parasitol 2019, 229, 15-23, doi:10.1016/j.molbiopara.2019.02.003.
- Casanova M, Portalès P, Blaineau C, Crobu L, Bastien P, Pagès M.(2008), Inhibition of active nuclear transport is an intrinsic trigger of programmed cell death in trypanosomatids. Cell Death Differ.15(12):1910-20. DOI: 10.1038/cdd.2008.132.
- Cautain B, Hill R, de Pedro N, Link W. (2015). Components and regulation of nuclear transport processes. FEBS J.282(3):445-62. DOI:10.1111/febs.13163.
- Chang CW, Couñago RL, Williams SJ, Bodén M, Kobe B. (2012). Crystal structure of rice importin-α and structural basis of its interaction with plant-specific nuclear localization signals. Plant Cell. 24(12):5074-88. DOI: 10.1105/tpc.112.104422.
- Chang CW, Williams SJ, Couñago RM, Kobe B.(2014). Structural basis of interaction of bipartite nuclear localization signal from Agrobacterium VirD2 with rice importin-α. Mol Plant. 7(6):1061-1064. DOI: 10.1093/mp/ssu014.
- Chook YM, Blobel (2001). Karyopherins and nuclear import. Curr Opin Struct Biol. 11(6):703-15. doi: 10.1016/s0959-440x(01)00264-0.
- Christy NCV, Petri Jr (2011). Mechanisms of adherence, cytotoxicity and phagocytosis modulate the pathogenesis of Entamoeba histolytica. Future Microbiol. 6(12):1501-19. doi: 10.2217/fmb.11.120.
- Cingolani, Petosa C., Weis K., Müller CW. (1999). Structure of importin-beta bound to the IBB domain of importin-alpha. Nature. 399(6733):221-9. doi: 10.1038/20367.
- Conti E, Kuriyan J. (2000). Crystallographic analysis of the specific yet versatile recognition of distinct nuclear localization signals by karyopherin alpha. Structure. 15;8(3):329-38. DOI:10.1016/s0969-2126(00)00107-6.
- Debnath, Parsonage D., Andrade RM, He C., Cobo ER, Hirata K., Chen S., García-Rivera G., Esther Orozco, Martínez MB., Gunatilleke SS., Barrios AM, Arkin MR, Leslie B Poole LB., McKerrow JH, Reed SL. (2012). A high-throughput drug screen for Entamoeba histolytica identifies a new lead and target. Nat Med. 18(6):956-60. doi: 10.1038/nm.2758.
- Dias SM, Wilson KF, Rojas KS, Ambrosio AL, Cerione RA. (2009). The molecular basis for the regulation of the cap-binding complex by the importins. Nat Struct Mol Biol.16(9):930-7. DOI: 10.1038/nsmb.1649.
- Fontes MR, Teh T, Jans D, Brinkworth RI, Kobe B. (2003). Structural basis for the specificity of bipartite nuclear localization sequence binding by importin-alpha. J Biol Chem. 278(30):27981-7. DOI:10.1074/jbc.M303275200.
- Fournier D, Palidwor GA, Shcherbinin S, Szengel A, Schaefer MH, Perez-Iratxeta C, Andrade-Navarro MA. (2013), Functional and genomic analyses of alpha-solenoid proteins. PLoS One. 8(11):e79894. DOI:10.1371/journal.pone.0079894.
- Goldfarb D., Corbett A., Mason D., Harreman M., Adam S. (2004). Importin a: a multipurpose nuclear-transport receptor. Trends Cell Biol.14 (9): 505-514. DOI:10.1016/j.tcb.2004.07.016.
- Görlich D, Panté N, Kutay U, Aebi U, Bischoff FR. (1996). Identification of different roles for RanGDP and RanGTP in nuclear protein import. EMBO J. 15(20):5584-94. DOI: https//doi.org/10.1002/j.1460-2075.1996.tb00943.x.
- Hanadate, Saito‐Nakano Y., Nakada Tsukui K, Nozaki T. (2016). Endoplasmic reticulum‐resident Rab8A GTPase is involved in phagocytosis in the protozoan parasite Entamoeba histolytica. Cell Microbiol.18(10): 1358–1373. doi: 10.1111/cmi.12570
- Haque, Huston CD, Hughes M, Houpt E, Petri Jr WA. (2003). Amebiasis. N Engl J Med. 348(16):1565-73. doi: 10.1056/NEJMra022710.
- Hoelz A, Blobel G. (2004). Cell biology: popping out of the nucleus. Nature.432(7019):815-6. DOI:10.1038/432815a
- Jovanovic-Talisman T, Tetenbaum-Novatt J, McKenney AS, Zilman A, Peters R, Rout MP, Chait BT. (2009), Artificial nanopores that mimic the transport selectivity of the nuclear pore complex. Nature.457(7232):1023-7. DOI: 10.1038/nature07600.
- Kimura, Nakamura T., Nawa Y. (2007), Experience with intravenous metronidazole to treat moderate-to-severe amebiasis in Japan, Am J Trop Med Hyg., 77(2):381-5.
- King MC, Lusk CP, Blobel G. (2006). Karyopherin-mediated import of integral inner nuclear membrane proteins. 442(7106):1003-7. DOI:10.1038/nature05075.
- Kobe B.(1999). Autoinhibition by an internal nuclear localization signal revealed by the crystal structure of mammalian importin alpha. Nat Struct Biol. 6(4):388-97. DOI:10.1038/7625.
- Kutay U, Bischoff FR, Kostka S, Kraft R, Görlich D. (1997). Export of importin alpha from the nucleus is mediated by a specific nuclear transport factor. Cell.90(6):1061-71. DOI:10.1016/s0092-8674(00)80372-4.
- Lam MH, Briggs LJ, Hu W, Martin TJ, Gillespie MT, Jans (1999). Importin beta recognizes parathyroid hormone-related protein with high affinity and mediates its nuclear import in the absence of importin alpha. J Biol Chem. 274(11):7391-8.doi: 10.1074/jbc.274.11.7391.
- Leung SW, Harreman MT, Hodel MR, Hodel AE, Corbett AH. (2003). Dissection of the karyopherin alpha nuclear localization signal (NLS)-binding groove: functional requirements for NLS binding. J Biol Chem. 278(43):41947-53. DOI:10.1074/jbc.M307162200.
- Lopez-Reyez I., Garcia-Rivera G., Banuelos C., Herranz S., Vincent O., Lopez-Camarillo C., Marchat LA., Orozco E. (2010). Detection of the endosomal sorting complex required for transport in Entamoeba histolytica and characterization of the EhVps4 protein. Journal of biomedicine and biotechnology. 890674
- Ma J, Goryaynov A, Yang W. (2016). Super-resolution 3D tomography of interactions and competition in the nuclear pore complex. Nat Struct Mol Biol. 23(3):239-47. DOI: 10.1038/nsmb.3174.
- Marie C and Petri Jr. WA (2013). Amoebic dysentery. BMJ Clin Evid. 2013:0918. PMID: 23991750
- Matunis MJ, Coutavas E, Blobel G. (1996). A novel ubiquitin-like modification modulates the partitioning of the Ran-GTPase-activating protein RanGAP1 between the cytosol and the nuclear pore complex. J Cell Biol.135(6 Pt 1):1457-70. DOI:10.1083/jcb.135.6.1457.
- Moore, Yang J., Truant R., Kornbluth S. (1999). Nuclear Import of Cdk/Cyclin Complexes: Identification of Distinct Mechanisms for Import of Cdk2/Cyclin E and Cdc2/Cyclin B1. J Cell Biol.144(2): 213–224. doi: 10.1083/jcb.144.2.213
- Moroianu J, Blobel G, Radu A. (1996). Nuclear protein import: Ran-GTP dissociates the karyopherin alphabeta heterodimer by displacing alpha from an overlapping binding site on beta. Proc Natl Acad Sci U S A. 93(14):7059-62. DOI:10.1073/pnas.93.14.7059.
- Oka M, Yoneda Y. (2018). Importin α: functions as a nuclear transport factor and beyond. Proc Jpn Acad Ser B Phys Biol Sci.94(7):259-274. DOI:10.2183/pjab.94.018.
- Pritt, Clark CG., (2008), Amebiasis, Mayo Clin Proc., 83(10):1154-9; quiz 1159-60.doi: 10.4065/83.10.1154.
- Ralston (2015).Chew on this: Amoebic trogocytosis and host cell killing by Entamoeba histolytica. 31(9): 442–452. doi: 10.1016/j.pt.2015.05.003
- Ralston KS., Solga MD, Mackey-Lawrence NM, Somlata Bhattacharya A., Petri Jr., WA (2014). Trogocytosis by Entamoeba histolytica contributes to cell killing and tissue invasion. Nature. 508 (7497), 526-530. doi: 10.1038/nature13242.
- Riggleman B, Wieschaus E, Schedl P.(1989). Molecular analysis of the armadillo locus: uniformly distributed transcripts and a protein with novel internal repeats are associated with a Drosophila segment polarity gene. Genes Dev.3(1):96-113. DOI:10.1101/gad.3.1.96.
- Ritterhoff T, Das H, Hofhaus G, Schröder RR, Flotho A, Melchior F. (2016). The RanBP2/RanGAP1*SUMO1/Ubc9 SUMO E3 ligase is a disassembly machine for Crm1-dependent nuclear export complexes. Nat Commun.7:11482. DOI: 10.1038/ncomms11482.
- Sahoo N., Labruyere E., Bhattacharya S., Sen P., Guillen N., Battacharya A. (2004). Calcium binding protein 1 of the protozoan parasite Entamoeba histolytica interacts with actin and is involved in cytoskeleton dynamics. Journal of Cell Science 117(Pt 16), 3625-3634.
- Saito-Nakano Y, Loftus BJ, Hall N, Nozaki (2005). The diversity of Rab GTPases in Entamoeba histolytica. Exp Parasitol. 110(3):244-52. doi: 10.1016/j.exppara.2005.02.021.
- Seewald MJ, Körner C, Wittinghofer A, Vetter IR. (2002). RanGAP mediates GTP hydrolysis without an arginine finger. Nature.415(6872):662-6. DOI:10.1038/415662a.
- Sha SM., Usmanghani K., Akhtar N., Akram M., Asif HM., Hasan MM., (2016). Clinical study on the efficacy of amoebex (coded herbal drug) compared with metronidazole for the treatment of amoebic dysentery. Pakistan Journal of Pharmaceutical Sciences 29(6), 2005-2014.
- Singh, Walia AK, Kanwar JR., (2016). Protozoa lectins and their role in host-pathogen interactions. 34(5):1018-1029.doi: 10.1016/j.biotechadv.2016.06.002.
- Somlata, Bhattacharya, Bhattacharya A., (2011)., A C2 domain protein kinase initiates phagocytosis in the protozoan parasite Entamoeba histolytica. Nat Commun. 2:230. doi: 10.1038/ncomms1199.
- Somlata, Kamanna, Agrahari M., Babuta M., Bhattacharya S., Bhattacharya A. (2012). Autophosphorylation of Ser428 of EhC2PK plays a critical role in regulating erythrophagocytosis in the parasite Entamoeba histolytica. J Biol Chem. 287(14):10844-52. doi: 10.1074/jbc.M111.308874.
- Stanley Jr, (2003), Amoebiasis, Lancet, 361(9362):1025-34. doi: 10.1016/S0140-6736(03)12830-9.
- Stewart M, Rhodes D. (1999). Switching affinities in nuclear trafficking. Nat Struct Biol.6(4):301-4. DOI:10.1038/7529.
- Stewart M. (2000). Insights into the molecular mechanism of nuclear trafficking using nuclear transport factor 2 (NTF2). Cell Struct Funct. 25(4):217-25. DOI:10.1247/csf.25.217.
- Stewart M. (2007). Molecular mechanism of the nuclear protein import cycle. Nat Rev Mol Cell Biol.8(3):195-208. DOI:10.1038/nrm2114
- Strambio-De-Castillia C, Niepel M, Rout MP. (2010). The nuclear pore complex: bridging nuclear transport and gene regulation. Nat Rev Mol Cell Biol.11(7):490-501. DOI: 10.1038/nrm2928.
- Verma, Nozaki T., Datta S. (2016). Role of EhRab7A in phagocytosis of type 1 fimbriated E. coli by Entamoeba histolytica. Mol Microbiol. 102(6):1043-1061. doi: 10.1111/mmi.13533.
- Verma, Saito-Nakano Y., Nozaki T., Datta S. (2015). Insights into endosomal maturation of human holo-transferrin in the enteric parasite Entamoeba histolytica: essential roles of Rab7A and Rab5 in biogenesis of giant early endocytic vacuoles. Cellular microbiology. 17(12), 1779-1796. doi.org/10.1111/cmi.12470
- Vetter IR, Nowak C, Nishimoto T, Kuhlmann J, Wittinghofer A. (1999). Structure of a Ran-binding domain complexed with Ran bound to a GTP analogue: implications for nuclear transport. 398(6722):39-46. DOI:10.1038/17969.
- Wagstaff KM, Sivakumaran H, Heaton SM, Harrich D, Jans DA. (2012). Ivermectin is a specific inhibitor of importin α/β-mediated nuclear import able to inhibit replication of HIV and dengue virus. Biochem J. 443(3):851-6. DOI: 10.1042/BJ20120150.
- Wente SR., Rout MP. (2010). The Nuclear Pore Complex and Nuclear Transport. Cold Spring Harb Perspect Biol.2(10): a000562. doi: 1101/cshperspect.a000562
- Wirthmueller L, Roth C, Fabro G, Caillaud MC, Rallapalli G, Asai S, Sklenar J, Jones AM, Wiermer M, Jones JD, Banfield MJ. (2015). Probing formation of cargo/importin-α transport complexes in plant cells using a pathogen effector. Plant J.81(1):40-52. DOI: 10.1111/tpj.12691.
- Xu L, Massagué J. (2004), Nucleocytoplasmic shuttling of signal transducers. Nat Rev Mol Cell Biol. 5(3):209-19. DOI:10.1038/nrm1331.
- Yang SN, Takeda AA, Fontes MR, Harris JM, Jans DA, Kobe B. (2010). Probing the specificity of binding to the major nuclear localization sequence-binding site of importin-alpha using oriented peptide library screening. J Biol Chem. 285(26):19935-46. DOI: 10.1074/jbc.M109.079574.
- Zulfiqar, Mathew G., Horrall S., (2023). Amebiasis. StatPearls Publishing.
Foto de portada: Entamoeba histolytica (Entamoeba histolytica) de Creative Commons por Doc. RNDr. Josef Reischig, CSc.