Un transcriptoma puede emplearse, en particular, para estudiar a las células troncales de un tejido. Ante esta afirmación, el lector puede preguntar ¿qué es un transcriptoma y cuál es su importancia? Por ello, este artículo tiene como objetivo explicar qué es un transcriptoma y cómo puede ser utilizado para caracterizar a las células que constituyen a los diferentes tejidos de nuestro cuerpo. En especial, nos enfocamos en el empleo de la transcriptómica para distinguir específicamente a las células troncales (células “madre”) del epitelio que recubre a la córnea; que es la región anterior del ojo y que, debido a su transparencia, se convierte en el principal sistema óptico del mismo. El estudio de los transcriptomas nos proporciona una herramienta muy poderosa, que nos permite analizar e identificar las diferentes funciones que tiene esta población celular en la renovación y reparación del tejido. A largo plazo, el entendimiento de cómo se regulan las células troncales puede tener repercusiones en medicina regenerativa y en el combate de algunas patologías del sistema ocular.
¿Qué es la transcriptómica?
Cuando observamos a un ser vivo, ya sea un animal, una planta, un hongo o una bacteria, lo que distinguimos son su tamaño o sus características morfológicas y funcionales. Estas cualidades son resultado de la actividad de una variedad de proteínas y otros componentes como los carbohidratos y lípidos, que estructuran, dan forma y movimiento, y confieren muchas otras propiedades inherentes al organismo que estudiamos. Así, cada organismo es el resultado de la actividad biológica de las proteínas que lo constituyen (Fig. 1).

El conocimiento actual ha acumulado evidencia de que las diferentes proteínas son producidas en los ribosomas libres que se localizan en el citoplasma celular, así como en los ribosomas asociados al retículo endoplásmico rugoso. Aquellas proteínas que se producen en el citoplasma permanecen en la célula y tienen funciones principalmente asociadas al metabolismo. En contraste, las proteínas que se producen en el retículo endoplásmico rugoso se insertan en las membranas celulares o son exportadas fuera de la célula, para llevar a cabo funciones como la comunicación célula-célula, como mensajeros que regulan el metabolismo del organismo, o bien como hormonas (Fig. 2).
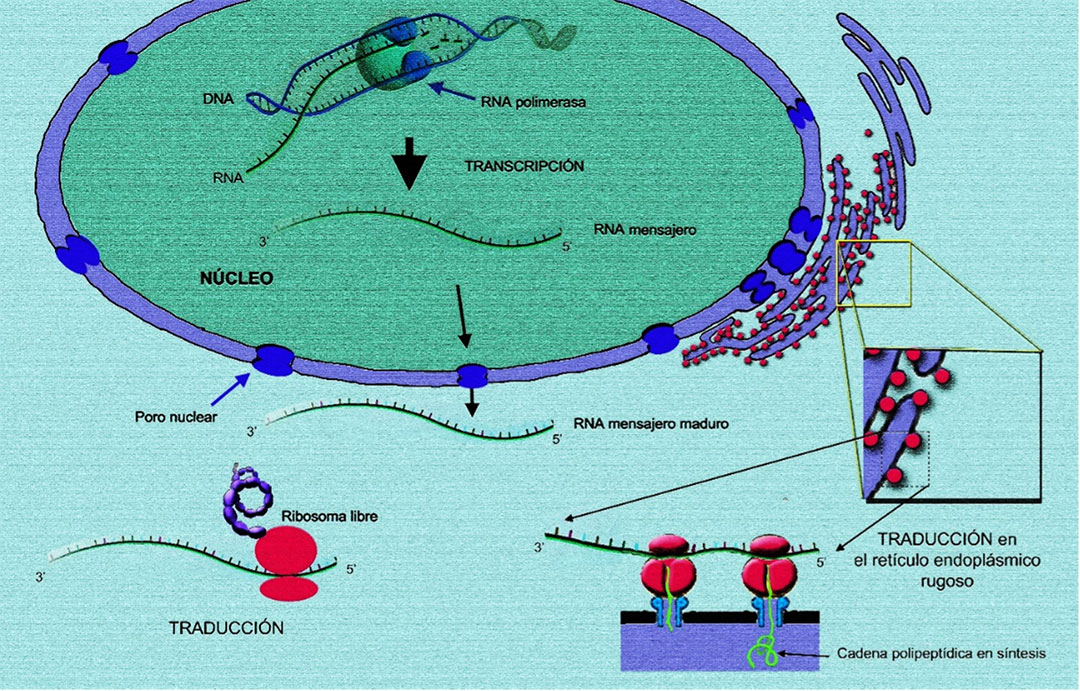
Las instrucciones que dictan la estructura y funcionalidad de las proteínas se encuentran en el genoma, que corresponde a todas las secuencias de ácidos nucleicos localizadas en el núcleo y que se conocen como genes. Para que las instrucciones contenidas en los genes puedan traducirse en proteínas, primero es necesario que la secuencia de un gene sea copiada en forma de RNA, proceso al que conocemos con el nombre de transcripción; y al proceso por el cual la información contenida en un gen se utiliza para dirigir la síntesis de una proteína funcional o de un ARN no codificante, se le conoce como expresión génica. Cabe aclarar que a cada RNA producido mediante el proceso de transcripción se le llama transcrito.
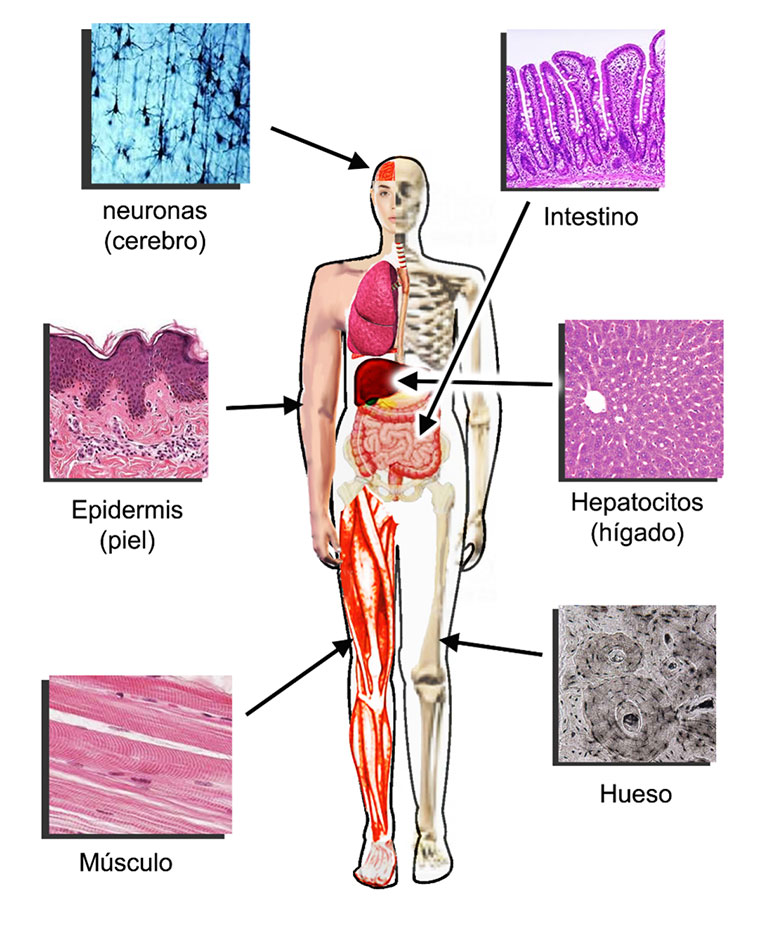
Si se piensa en los diferentes tejidos que componen los órganos del cuerpo, cada uno de ellos expresa una colección de genes que determinan sus funciones que en el organismo. De este modo, las células que componen a la epidermis, al hígado, a los huesos, al intestino, o las neuronas de nuestro cerebro, expresan diferentes colecciones de transcritos que codifican a las proteínas que les dan sus funciones (Fig. 3). A cada una de estas colecciones de transcritos específicos para cada tejido, las conocemos con el nombre de transcriptoma.
Es importante señalar que el transcriptoma de un tejido puede variar durante el desarrollo de un individuo, o bien, tanto en respuesta a los estímulos que el tejido recibe de su entorno o debido a las alteraciones ocasionadas por un daño o un patógeno. Como ejemplo podemos mencionar los cambios que ocurren en las células de la epidermis cuando se desarrolla el cáncer de piel (carcinoma escamoso). Si comparamos el transcriptoma de células normales con el de células de un carcinoma escamoso, encontraremos cambios sustanciales que llevan al crecimiento tumoral descontrolado y a la pérdida de características típicas de la epidermis.
¿Por qué es importante conocer el transcriptoma de una célula o un tejido?
La transcriptómica o análisis de los transcriptomas completos, abarca a un conjunto de tecnologías que tienen como propósito estudiar a todos los transcritos que se expresan en una célula o tejido en un momento determinado. En otras palabras, la transcriptómica es el análisis y la caracterización de todas las moléculas de RNA, tanto codificantes como no codificantes, contenidas en una población celular o bien en una sola célula.
Puesto que no todos los tejidos responden de la misma manera a estímulos como pueden ser las hormonas, o bien, cambian sus características durante las diferentes etapas del desarrollo de un individuo o de una enfermedad, la idea de identificar a todos los RNAs contenidos en una célula, parte de la necesidad de conocer y comparar la expresión de genes entre poblaciones de células, para entender los mecanismos que conducen a estos cambios, así como los procesos que intervienen en la reparación de los tejidos o en el desarrollo de enfermedades como el cáncer.
¿Cómo se obtiene un transcriptoma?
Un transcriptoma se obtiene mediante la secuenciación, identificación y cuantificación del conjunto de moléculas de RNA presentes en una muestra biológica. Este proceso permite estudiar qué genes se están expresando y en qué niveles se encuentran. Puesto que el transcriptoma es específico de cada tejido, los estudios realizados en muestras derivadas de un tejido particular no necesariamente representan el perfil de expresión en un tejido diferente. Por ello, al comparar el transcriptoma de células de la epidermis con el transcriptoma del tejido óseo, es de esperarse que existan diferencias notables entre cada uno de ellos; lo mismo sucede al comparar un tejido normal, con una muestra de tejido neoplásico derivado del mismo.
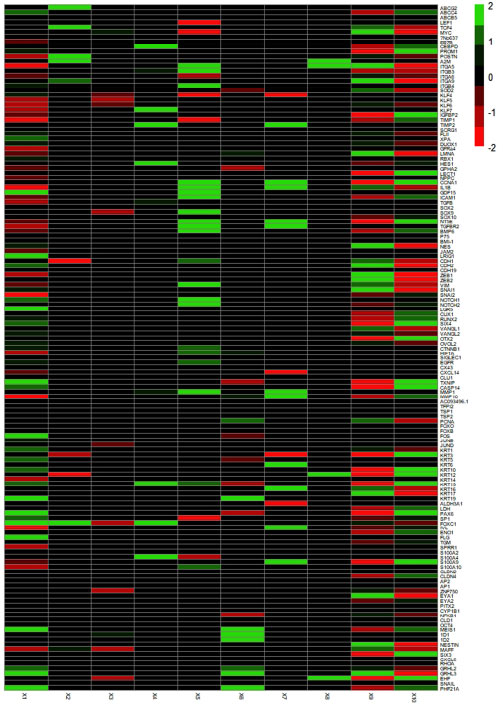
Para hacer el análisis comparativo entre diferentes transcriptomas, una vez secuenciado el RNA de la muestra analizada, se recurre a una serie de herramientas bioinformáticas que permiten identificar y cuantificar los transcritos correspondientes, al mismo tiempo que se establecen los parámetros que definen si los cambios detectados son significativos. Una de las formas de identificar las similitudes y diferencias en la expresión genética al comparar muestras de diferentes tejidos o etapas de desarrollo, consiste en la elaboración de mapas de calor o “heatmaps”. Éstos son representaciones visuales de los datos de expresión génica, utilizando colores para mostrar la intensidad de la expresión de diferentes genes en diversas condiciones o muestras (Fig. 4). Para elaborarlos, los datos se organizan en una tabla donde las filas corresponden a cada uno de los genes que componen a las muestras, y las columnas corresponden a las muestras o condiciones que se comparan. Una vez que se elabora esta tabla, a cada celda se le asigna un color basado en la intensidad de la expresión génica. Se asignan colores cálidos (como rojo o naranja) para indicar niveles altos en la expresión de los genes, mientras que la baja expresión se asocia a colores fríos (como el azul y el verde). No obstante, la asignación de colores puede hacerse con base en otros criterios, por lo que los investigadores siempre acotan cómo se hizo tanto la asignación de los colores, así como los niveles mínimos para que los resultados sean considerados como significativos (Fig.4).
Análisis comparativo de los transcriptomas reportados para las poblaciones de células “madre” (células troncales) de la superficie ocular.
Uno de los aspectos de interés para nuestro grupo de investigación consiste en estudiar la biología del epitelio que recubre la porción transparente del ojo de los mamíferos: la córnea. Esto debido a que, para este epitelio (epitelio corneal), se propuso que la población de células troncales que interviene en la reparación y renovación de la superficie ocular se encuentra en la región anatómica conocida como limbo esclero-corneal (Fig. 5).
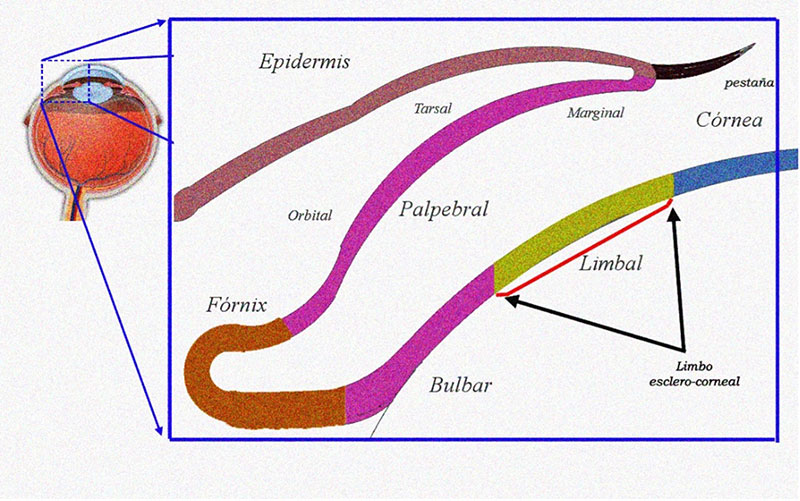
¿Por qué es importante la presencia de células troncales en la superficie ocular? El epitelio corneal se caracteriza por ser un epitelio estratificado, constituido por 5-10 capas de células (Fig. 6).
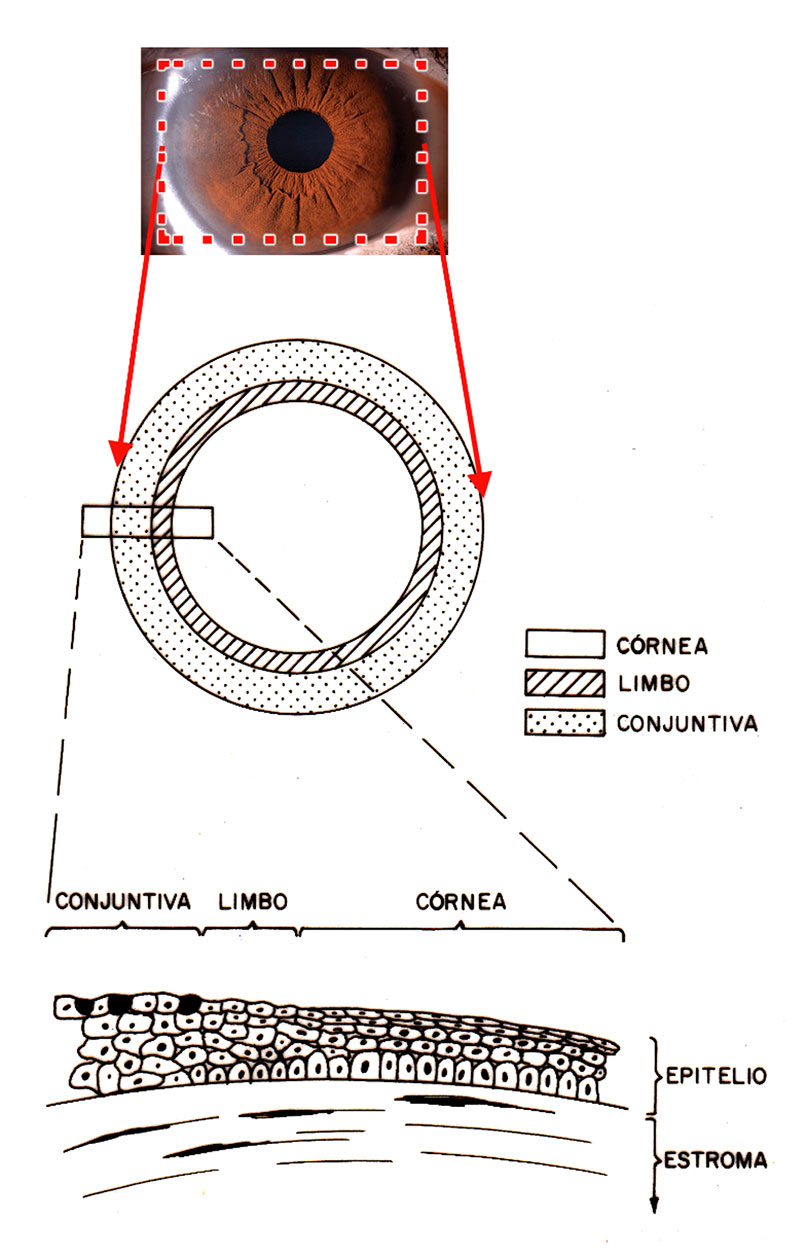
Estas se encuentran en renovación continua, debido a que las células de las capas superiores se descaman, de manera similar a como ocurre en la epidermis. Por ello, las células troncales del epitelio corneal tienen gran relevancia, debido a que participan en la renovación normal del tejido, así como también intervienen en los procesos de regeneración del tejido subsecuentes a una lesión, como las que ocurren por el mal uso de los lentes de contacto, o las que se presentan después de infecciones con virus como herpes zoster (Fig. 7).
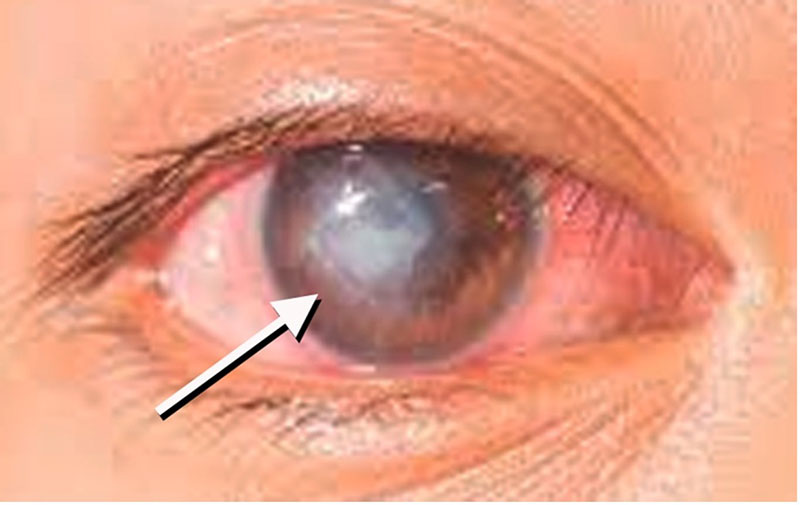
Considerando la importancia de esta población, es necesario conocer cuáles son los mecanismos que la regulan; sobre todo, entender los procesos que determinan la capacidad de estas células para multiplicarse y migrar en respuesta a las agresiones del medio ambiente, para regenerar al tejido una vez que ocurre una lesión.
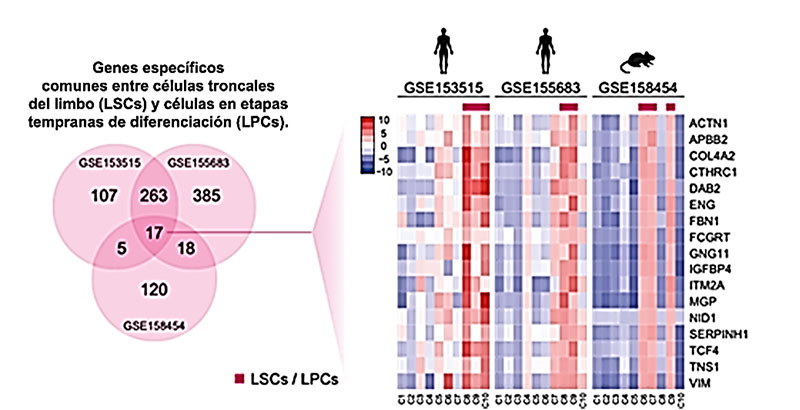
Considerando que hasta ahora no se han descrito moléculas que permitan distinguir de manera específica a las células troncales corneales del resto de las células que constituyen al tejido epitelial, hemos propuesto hacer un análisis comparativo de los transcriptomas reportados para las células del limbo esclero-corneal, obtenidos de diferentes especies de mamíferos, y mediante estrategias metodológicas distintas. Para hacer este análisis, también consideramos a una línea celular que in vitro, es decir en el laboratorio, reproduce el proceso de diferenciación de epitelio corneal.
A la fecha, hemos hecho la comparación entre los transcriptomas de células del limbo esclero-corneal de humano, y de ratón (Fig. 8). Hasta ahora los resultados permitieron identificar algunos marcadores de interés, aunque todavía no podemos asegurar que se trata de moléculas que determinan las características distintivas de la población de células troncales del epitelio corneal. Esta variabilidad se relaciona con las diferencias entre las especies de origen del RNA utilizado para el análisis, con el uso de metodologías distintas, y con diferencias en las edades de los organismos utilizados como fuente del tejido. Asimismo, no podemos descartar el hecho de que el limbo esclero-corneal está constituido por una mezcla heterogénea de células que se encuentran en diferentes estados de diferenciación (o especialización).
¿Cuál es la utilidad de este análisis?
Aunque recientemente se ha profundizado en el conocimiento de algunos de los factores que regulan la expresión del proceso de diferenciación terminal del epitelio corneal, aún no ha sido posible identificar a las moléculas que puedan ser utilizadas por los investigadores para aislar y caracterizar a las células troncales de este tejido.
Por ello, se hace necesaria la identificación de marcadores moleculares que permitan localizar con mayor precisión a las células troncales epiteliales y definir si existen otros sitios de la superficie ocular donde se puedan albergar.
De manera adicional, se hace necesaria la identificación de marcadores moleculares específicos de las células troncales de epitelio corneal, ya que su localización y caracterización molecular permitirá su uso para mejorar la regeneración corneal cuando la superficie ocular sufre una destrucción masiva como la que ocurre con quemaduras, o bien, para desarrollar tratamientos de patologías donde el limbo esclero-corneal es deficiente como en el pterigión (es decir, las carnosidades que se observan en algunos pacientes), o como la terapia posterior a las deficiencias asociadas a la cirugía por glaucoma.
Lecturas recomendadas
- Castro-Muñozledo, F. (2025). Análisis de perfiles transcripcionales: ventajas y dificultades en su ejecución. Revista de Educación Bioquímica 44(2):82-98.
- Ortiz-Melo, M.T.; Campos, J.E.; Sánchez-Guzmán, E.; Herrera-Aguirre, M.E.; Castro-Muñozledo, F. (2024). Regulation of corneal epithelial differentiation: miR-141-3p promotes the arrest of cell proliferation and enhances the expression of terminal phenotype. PLoS ONE. 19(12): e0315296. (doi: 10.1371/journal.pone.0315296).
- Ortiz-Melo, M.T.; García-Murillo, M.J.; Salazar-Rojas, V.M.; Campos, J.E.; Castro-Muñozledo, F. (2021). Transcriptional Profiles along Cell Programming into Corneal Epithelial Differentiation. Exp. Eye Res. 202:108302. (doi: 10.1016/j.exer.2020.108302).
- Castro-Muñozledo, F. (2015). The Mammalian Limbal Stem Cell Niche: A Complex Interaction between Cells, Growth Factors and Extracellular Matrix. In: Biology in Stem Cell Niche, Chapter 2. Stem Cell Biology and Regenerative Medicine Book Series. (Turksen, K., editor). E-Book ISBN: 978-3-319-21702-4; Hardcover ISBN: 978-3-319-21701-7. Series ISSN: 2196-8985. Springer International Publishing Switzerland. (doi: 10.1007/978-3-319-21702-4_2). Pages 23-56.