En este año el Premio Nobel de Fisiología y Medicina se otorgó a los científicos Víctor Ambros y Gary Ruvkun por un descubrimiento que transformó nuestra comprensión de cómo se activan y desactivan los genes. Este hallazgo revolucionó la investigación clínica por sus aplicaciones potenciales en el diagnóstico y tratamiento del cáncer, las afecciones neurodegenerativas y las enfermedades autoinmunes.
Introducción
Toda la información que determina cómo somos se encuentra guardada en nuestros genes contenidos en los cromosomas, que actúan como un manual de instrucciones para todas las células de nuestro cuerpo. Aunque cada célula contiene la misma información, los distintos tipos celulares presentan características y funciones únicas. Esto es, las células epiteliales forman una capa de piel y recubren los órganos, cumpliendo funciones protectoras y de absorción, mientras que las sanguíneas transportan el oxígeno y defienden al cuerpo contra las infecciones. Esta especialización de las células, que les permite responder a diferentes condiciones, surge de la regulación génica, proceso mediante el cual se controla la expresión de los genes, determinando cuáles se activan o desactivan en un momento dado.
Los galardonados Víctor Ambros y Gary Ruvkun, interesados en cómo se desarrollan los distintos tipos celulares, descubrieron una clase de ARNs pequeños, denominados micro ARNs (miARNs), cruciales en la regulación de la expresión genética. Al igual que el descubrimiento del ADN como portador de la información genética en los años 50, el hallazgo de los miARNs ha marcado un antes y un después en la biología molecular, al revelar una nueva y revolucionaria forma de control genético.
El Dogma Central de la Biología Molecular.
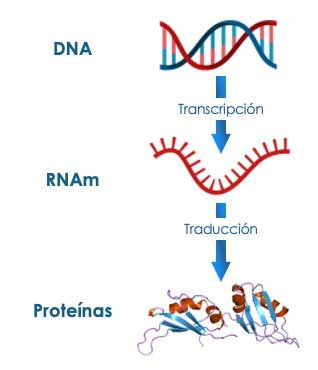
La manera en que la célula utiliza la información genética y la transmite a las células hijas para mantener la vida y permitir el desarrollo, se conoce como Dogma Central de la Biología Molecular, propuesto por uno de los galardonados con el Premio Nobel de Medicina de 1962, Francis Crick, y que establece que la información genética en las células fluye en una sola dirección y en tres pasos:
1) Replicación: el ADN se copia para producir nuevas moléculas de ADN, con lo que se asegura que este almacén de información genética, se transmita a las células hijas durante la división celular.
2) Transcripción: el ADN se copia a un ARN mensajero (ARNm) que sirve como intermediario temporal, que descifra la información contenida en el ADN.
3) Traducción: la información contenida en este ARNm se traduce en proteínas, que son las moléculas encargadas de ejecutar las funciones esenciales en la célula, y responsables de la mayoría de los procesos biológicos (Fig. 1).
Si bien el Dogma Central nos explica cómo se producen las proteínas, no establece el cuándo y el cómo se regula la expresión de las proteínas.
Mecanismos de regulación génica: cómo las células controlan la expresión de sus genes.
Dado que la información genética está contenida en todas las células de cada organismo, siempre ha existido gran interés en entender cómo éstas aseguran que se produzcan las proteínas correctas en el momento adecuado. Ello se logra a través de la regulación de la expresión génica, que controla cuándo, dónde y cuánto expresa o no un determinado gen. Para la fecha en que Ambros y Ruvkun publican sus trabajos, ya se conocían algunos de estos mecanismos para controlar la activación y desactivación de genes; éstos eran:
- Regulación transcripcional: este mecanismo controla la producción de ARN ante determinados estímulos, lo que ocurre cuando ciertos factores proteicos se unen a secuencias específicas del ADN para promover o inhibir la producción de un RNAm.
- Regulación post-transcripcional: después de que el ARN ha sido producido, se lleva a cabo una serie de modificaciones que permiten modular su estabilidad o vida media, afectando la cantidad de la proteína que se producirá.
- Regulación traduccional: aquí se controla cuánta proteína se produce a partir de un ARNm, la producción de diferentes variantes de una misma proteína, o cuánto tiempo debe usarse el ARN antes de ser degradado.
- Regulación post-traduccional: una vez que se ha producido la proteína, esta regulación se encarga de modificarla, con lo cual se altera su función o estabilidad.
- Regulación epigenética: este tipo de regulación no altera la secuencia del ADN, pero sí modifica su estructura o las proteínas asociadas a él, lo que puede afectar cómo se expresan los genes a largo plazo.
Cada uno de estos mecanismos trabaja en conjunto para que las células produzcan las proteínas correctas en las cantidades adecuadas y en el momento preciso. Sin embargo, el descubrimiento de los miARNs cambió radicalmente nuestra concepción de los mecanismos de regulación de la expresión génica, al revelar un proceso desconocido previamente, que afecta casi todos los aspectos de la biología celular. Estos pequeños miARNs actúan como “interruptores” que activan y desactivan genes cruciales para el desarrollo y la salud.
Los descubrimientos clave detrás de la regulación de los genes por los microARNs.
A principios de los años 90, los investigadores Ambros y Ruvkun estudiaban los procesos biológicos fundamentales, como el desarrollo y diferenciación celular utilizando el gusano nematodo Caenorhabditis elegans (C. elegans). Este pequeño gusano, que se ha vinculado a descubrimientos generadores del Premio Nobel en varias ocasiones, es un modelo que se ha utilizado ampliamente en investigación debido a características como tener un ciclo de vida corto (de aproximadamente 3 días), un número fijo de células especializadas (cerca de 1000 células somáticas de adulto), y ser fácil de cultivar y manipular genéticamente, lo que ha permitido investigar funciones genéticas específicas. Además, su cuerpo es transparente, por lo que podemos observar fácilmente el impacto de estas funciones en el desarrollo celular y los procesos internos en tiempo real con un microscopio.
Ambos investigadores, trabajando en conjunto pero de manera independiente, se enfocaron en comprender cómo los genes controlan la temporalidad de la diferenciación celular y encontraron dos genes clave de este proceso: lin-14 y lin-4. El gen lin-14 codifica a la proteína reguladora del desarrollo LIN-14, que marca el ritmo de los eventos celulares que se requieren para que el gusano C. elegans se vaya desarrollando. Esta proteína es un factor de la transcripción que, cuando está presente en niveles altos, permite que las células permanezcan en una etapa temprana del desarrollo. A medida que su nivel disminuye, las células pueden avanzar a etapas posteriores del desarrollo larval. En otras palabras, la proteína LIN-14 influye en los patrones de división celular y el destino de las células. El gen lin-14 es parte de una ruta genética que incluye a otros genes reguladores de tiempo, como lin-4, que, a pesar de transcribirse a un ARN, no es traducido en una proteína.
Pero ¿cómo se relacionan estos genes? La respuesta está en una historia que derivó en el descubrimiento de un proceso fundamental en la biología molecular y abrió el camino para entender un nuevo modelo de regulación génica: el mediado por los pequeños ARNs no codificantes.
El sorprendente descubrimiento de los microARNs: clave para descifrar los secretos de la regulación génica.
Los científicos galardonados, Ambros y Ruvkun, estudiaron dos cepas mutantes de C. elegans, que mostraban defectos evidentes en su crecimiento y desarrollo con el objetivo de identificar los genes mutados y entender cuál era la función que regulaban. Ambros había determinado que gen lin-4 actuaba como un regulador negativo del gen lin-14, pero el mecanismo a través del cual ocurría ésto aún era desconocido.
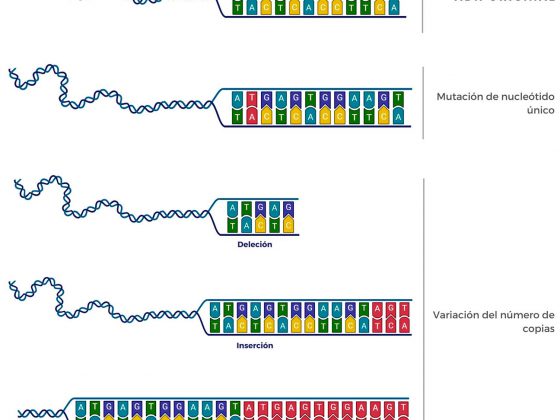
Ambos investigadores descubrieron que el transcrito del gen lin-4 era un miARN de apenas 22 nucleótidos (nt) de longitud, que no codificaba para alguna proteína. Sin embargo, su secuencia era complementaria a una región específica en el extremo 3’ no codificante del ARNm de lin-14, conocida como región no traducida 3’ (RNT 3’), (Fig. 2). La interacción de ambos ARNs resultaba en la inhibición de la síntesis de la proteína LIN-14, crucial para el desarrollo de este gusano.
El descubrimiento de Ambros y Ruvkun sentó las bases de un nuevo mecanismo de regulación génica, fundamental para el funcionamiento de los tejidos y el desarrollo de los organismos. Pero ¿cómo ocurre este control de la expresión génica?
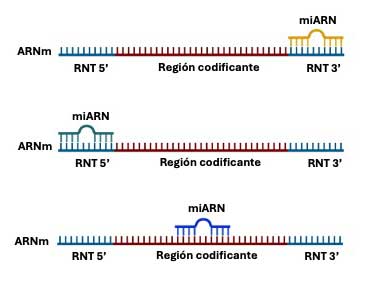
Los microARNs en la regulación de los genes.
Los miARNs son pequeñas moléculas de ARN no codificantes, de entre 21 y 24 nt de longitud que, al unirse a secuencias complementarias presentes en los ARNm, suelen derivar en la degradación de dicho ARNm, controlando así su expresión.
Los genes a partir de los cuales se generan los miARNs, pueden estar en diferentes partes del ADN. Algunos se encuentran entre regiones codificantes de proteínas (regiones intergénicas); otros se ubican dentro de regiones que se editan durante la maduración de los ARNs, conocidas como intrones y otros en exones no codificantes, que son partes de genes que no producen proteínas, pero que tienen otras funciones en la célula.
El proceso de transcripción y maduración de los miARNs comienza en el núcleo celular donde se transcriben principalmente por acción de la ARN polimerasa II (la misma polimerasa que transcribe los ARNm), como precursores largos llamados pri-mARNs, que son procesados por la Ribonucleasa (ARNsa) Drosha, una enzima que degrada al ARN en regiones específicas de la base de una estructura de tallo y burbuja, y produce ARNs precursores cortos de doble cadena, llamados pre-miARNs (Fig. 3). Estos ARNs se exportan del núcleo al citoplasma mediante su interacción con el complejo proteico exportina 5/RanGTP. Una vez en el citoplasma, otra enzima llamada Dicer, reconoce y procesa la burbuja terminal del pre-ARN para producir un miARN de doble cadena de entre 21 y 24 nt de longitud. Finalmente, este dúplex se separa y cualquiera de las dos cadenas se une a un complejo de silenciamiento denominado RISC (por sus siglas en inglés ARN-induced silencing complex, o complejo de silenciamiento inducido por ARN), que interacciona con los ARNm blanco por complementariedad entre las secuencias de ambos ARNs.
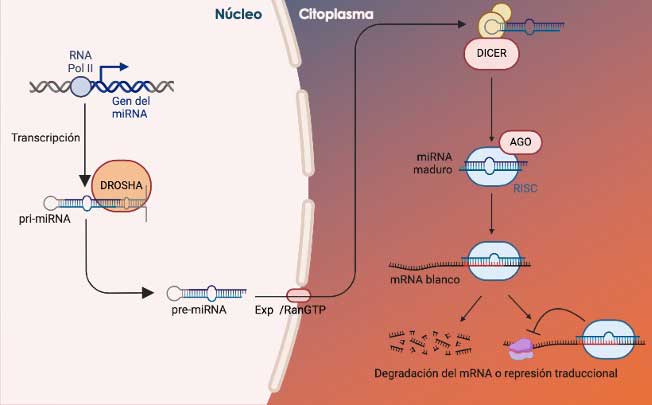
Finalmente, la proteína Argonauta (AGO) que es parte de este complejo, es la encargada de degradar el ARN blanco. Si bien los complejos de proteínas llevan a cabo la degradación de los mARNs, los miRNAs son los que actúan como controles maestros de este mecanismo para escoger al ARNm indicado.
En general, el principal sitio de interacción de los miARNs con su ARNm blanco es la RNT 3’, aunque también pueden hacerlo en las RNT 5’ y en raras ocasiones en las regiones codificantes (Fig. 2). Estas interacciones pueden bloquear la síntesis de la proteína o inducir la degradación de su ARNm, con lo que se impide su producción (Fig. 3) (O’Brien et al., 2018). Esta vía de síntesis de los miARNs se conoce como canónica. Su mecanismo de acción constituye una manera de regulación altamente especializada de muchos genes implicados en procesos clave como el desarrollo, la diferenciación celular, la homeostasis y la respuesta a diferentes estímulos y estrés.
Los microARNs, claves en la regulación génica y su impacto en la salud.
La capacidad de los miARNs para regular la expresión génica depende de factores como su cantidad, ubicación dentro de la célula, tipo celular y estado en el que se encuentren en un momento dado. Curiosamente, se ha estimado que, en los diferentes tipos celulares, el 50% de los miARNs, es decir, los más abundantes, corresponden a tan solo 5 de estas pequeñas moléculas; otro 25% está compuesto por miARNs que se expresan en todas las células, y el 25% restante corresponde a miARNs que se expresan en niveles muy bajos, con independencia del tipo celular (de Rie et al., 2017).
Como las funciones regulatorias de los miARNs son tan importantes en el crecimiento, desarrollo y homeostasis de los organismos, cualquier alteración en su actividad puede resultar en estrés celular o la aparición de diversas enfermedades. En las células sanas, los miARNs actúan como reguladores que ayudan a estabilizar las variaciones inesperadas en la actividad de los genes. Sin embargo, cuando su expresión se altera, ya sea porque hay demasiado o muy poco de ellos, se pueden generar desequilibrios en la cantidad de las proteínas que regulan. Esto desencadena problemas en las vías celulares en las que participan y, eventualmente, contribuyen al desarrollo de enfermedades. Estudiar el origen de estos cambios en los niveles y localización de los miARNs podría ofrecer nuevas oportunidades para restaurar su equilibrio de manera oportuna, lo que ayudaría a prevenir enfermedades (Seyhan, 2024).
Estudios realizados tanto en cultivos celulares como en modelos animales, en los que se ha demostrado una relación entre los miARNs y diversas enfermedades, han sido fundamentales para sentar las bases de ensayos clínicos que buscan establecer la seguridad y eficacia de las terapias basadas en miARNs, especialmente en el tratamiento de cáncer, enfermedades neurodegenerativas, cardiacas e infecciosas.
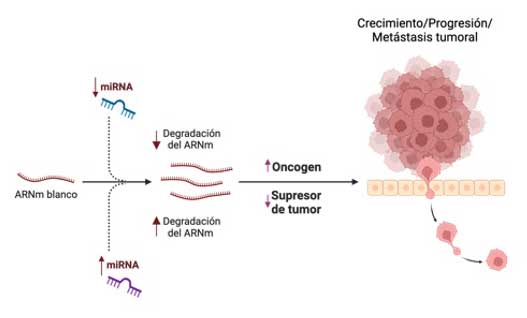
Los microARNs: inhibidores y promotores del cáncer.
En el ámbito del cáncer, se ha comprobado que algunos miARNs actúan como supresores de tumores, ayudando a inhibir el crecimiento celular descontrolado. Entre estos se encuentran let-7a/b, miR-16, miR-29b, miR-34a, miR-100, miR-122, miR-133b, miR-204-5p, miR-379 y miR-634 (O’Neill & Dwyer, 2020). Por otro lado, existen miARNs que funcionan como oncogenes, es decir, promueven el desarrollo de tumores cuando se expresan en niveles anormalmente altos (Fig. 4). Algunos ejemplos de estos son el miR-9, miR-10b, miR-17, miR-21, miR-132, miR-155, miR-222 y miR-375 (Fu et al., 2021). Restaurar los niveles naturales de expresión de miARNs puede representar una estrategia eficaz contra el cáncer, ya que su desequilibrio se relaciona con el desarrollo de la enfermedad. La regulación terapéutica de los miARNs ofrece un enfoque prometedor para tratar múltiples tipos de cáncer, así como otras enfermedades neurodegenerativas y cardiacas.
Terapias basadas en miARNs: nueva esperanza en el tratamiento de enfermedades.
Las terapias basadas en miARNs buscan regular la actividad de estas pequeñas moléculas cuando su equilibrio está alterado. Un ejemplo es el miR-155, que desempeña un papel importante en la respuesta del sistema inmunológico. Se ha observado que los niveles de miR-155 están elevados en pacientes con linfoma, un tipo de cáncer que afecta al sistema linfático. Para contrarrestar este desequilibrio, se llevan a cabo ensayos clínicos en los que se utilizan moléculas modificadas que actúan como inhibidores de este miARN. Los resultados de la primera fase de estos ensayos han sido prometedores: el tratamiento fue bien tolerado por los pacientes y mejoró su calidad de vida (Anastasiadou et al., 2021; Seyhan, 2024).
Además, esta misma estrategia terapéutica se está aplicando a otras enfermedades autoinmunes, como la artritis reumatoide, el lupus, la esclerosis múltiple y el síndrome de Sjögren, en las cuales también se ha detectado un aumento de los niveles del miR-155 (Xu et al., 2022).
Los miARNs también han sido investigados en el tratamiento de tumores sólidos, como el melanoma, el carcinoma renal y el hepatocarcinoma, en los que se ha detectado niveles reducidos del miR-34a. Este miARN actúa como un supresor de tumores, ya que su función se relaciona con la inhibición de procesos clave para el cáncer, como la proliferación, migración, invasión y metástasis de las células tumorales. El tratamiento consiste en la inyección intravenosa de nanopartículas liposomales, que transportan una versión sintética de miARN-34a, diseñada para restaurar su función. Aunque los estudios preclínicos (en modelos animales) mostraron resultados prometedores, los estudios clínicos en humanos no fueron exitosos, debido a la aparición de efectos adversos graves, algunos de los cuales resultaron fatales (Hong et al., 2020).
Terapias basadas en microARNs: desafíos y avances.
Uno de los principales desafíos que enfrentan las terapias basadas en miARNs, es asegurar que los inhibidores o mimetizadores de estos miARNs lleguen de manera eficiente a las células específicas donde se necesita el tratamiento. Esto es crucial para garantizar que la terapia actúe en el tejido adecuado, evitando efectos tóxicos y posibles alteraciones no deseadas en otras células o vías que no son el objetivo de la terapia.
Además, es fundamental encontrar estrategias que prolonguen la vida media de estas pequeñas moléculas en el organismo, de modo que puedan mantenerse activas el tiempo suficiente para regular la expresión de los genes en las células afectadas. Afortunadamente, en los últimos años se han logrado grandes avances en este campo. El uso de la nanotecnología, de adenovirus oncolíticos (virus modificados que atacan células tumorales), y diversas técnicas de modificación de ácidos nucleicos, han mejorado considerablemente los resultados en estudios preclínicos y clínicos. Considerando estos avances, se espera que en el futuro cercano las terapias basadas en miARNs se conviertan en una herramienta confiable y eficaz dentro de la medicina moderna.
Los microARNs: claves para detectar enfermedades.
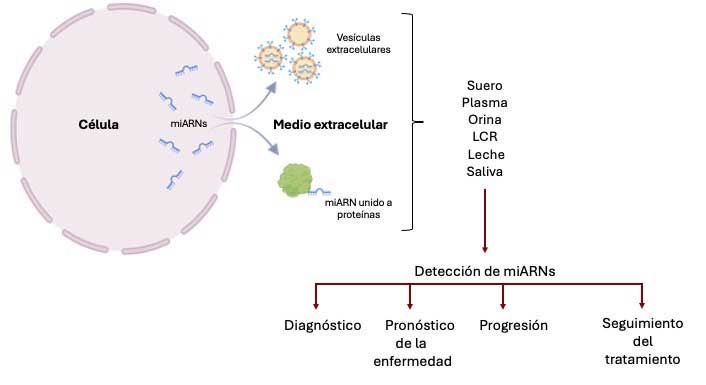
Los miARNs pueden encontrarse en fluidos extracelulares como el suero, el plasma, la orina, el líquido cefalorraquídeo (LCR), la saliva y la leche, ya sea en forma libre o dentro de pequeñas vesículas originadas de las células. Esto les permite establecer comunicación tanto con células vecinas como con células en otros tejidos.
La presencia de los miARNs en el ambiente extracelular, su estabilidad y el hecho de que su abundancia cambie significativamente en diversas enfermedades, los convierte en excelentes biomarcadores.
Un biomarcador es una molécula que permite identificar una enfermedad o condición incluso antes de que aparezcan los síntomas, lo que resulta crucial para un mejor manejo de la enfermedad. Además, su detección puede ayudar a determinar la progresión de una enfermedad y a dar seguimiento a la efectividad del tratamiento en curso.
Actualmente, se llevan a cabo protocolos clínicos que exploran el uso de miARNs como biomarcadores sensibles y específicos de enfermedades infecciosas, neurodegenerativas, cardiovasculares y cáncer. Debido a su presencia en el medio extracelular, los miARNs podrían detectarse mediante procedimientos poco invasivos. Por ejemplo, el miR-21-5p, ha sido identificado como un biomarcador potencial para el diagnóstico de cáncer de mama, pulmón, de colon, hueso, próstata y leucemia, debido a su rol en la promoción de la sobrevivencia celular y la metástasis tumoral (Nemeth et al., 2024). Por otro lado, cambios en los niveles séricos de los miR-17, miR-202 y miR-106b podrían utilizarse como indicadores de función cardíaca e hipertensión (Shah et al., 2018).
Estas pequeñas moléculas también podrían ser útiles en el diagnóstico temprano y el pronóstico de enfermedades neurodegenerativas, como la esclerosis lateral amiotrófica (ELA), una patología que afecta a las neuronas motoras y provoca parálisis progresiva. La detección precoz de miARNs como el miR-181 ofrecería la posibilidad de iniciar tratamientos oportunos que mejoren la calidad de vida de los pacientes (Magen et al., 2021).
Un futuro prometedor.
Todo este avance permite comprender por qué Víctor Ambros y Gary Ruvkun fueron galardonados merecidamente con el Premio Nobel de Fisiología y Medicina 2024. Su descubrimiento no solo amplió nuestra comprensión sobre la regulación de la expresión génica, sino que continúa siendo una fuente inagotable de innovación para tratamientos de medicina personalizada.
En el futuro cercano, los miARNs desempeñarán un papel clave en el tratamiento y manejo de enfermedades complejas, desde el cáncer hasta patologías autoinmunes, ofreciendo nuevas esperanzas en el campo de la medicina.
Referencias
Anastasiadou, E., Seto, A. G., Beatty, X., Hermreck, M., Gilles, M. E., Stroopinsky, D., Pinter-Brown, L. C., Pestano, L., Marchese, C., Avigan, D., Trivedi, P., Escolar, D. M., Jackson, A. L., & Slack, F. J. (2021, Feb 15). Cobomarsen, an Oligonucleotide Inhibitor of miR-155, Slows DLBCL Tumor Cell Growth In Vitro and In Vivo. Clinical Cancer Research, 27(4), 1139-1149. https://doi.org/10.1158/1078-0432.CCR-20-3139
de Rie, D., Abugessaisa, I., Alam, T., Arner, E., Arner, P., Ashoor, H., Astrom, G., Babina, M., Bertin, N., Burroughs, A. M., Carlisle, A. J., Daub, C. O., Detmar, M., Deviatiiarov, R., Fort, A., Gebhard, C., Goldowitz, D., Guhl, S., Ha, T. J., Harshbarger, J., Hasegawa, A., Hashimoto, K., Herlyn, M., Heutink, P., Hitchens, K. J., Hon, C. C., Huang, E., Ishizu, Y., Kai, C., Kasukawa, T., Klinken, P., Lassmann, T., Lecellier, C. H., Lee, W., Lizio, M., Makeev, V., Mathelier, A., Medvedeva, Y. A., Mejhert, N., Mungall, C. J., Noma, S., Ohshima, M., Okada-Hatakeyama, M., Persson, H., Rizzu, P., Roudnicky, F., Saetrom, P., Sato, H., Severin, J., Shin, J. W., Swoboda, R. K., Tarui, H., Toyoda, H., Vitting-Seerup, K., Winteringham, L., Yamaguchi, Y., Yasuzawa, K., Yoneda, M., Yumoto, N., Zabierowski, S., Zhang, P. G., Wells, C. A., Summers, K. M., Kawaji, H., Sandelin, A., Rehli, M., Consortium, F., Hayashizaki, Y., Carninci, P., Forrest, A. R. R., & de Hoon, M. J. L. (2017, Sep). An integrated expression atlas of miRNAs and their promoters in human and mouse. Nature Biotechnology, 35(9), 872-878. https://doi.org/10.1038/nbt.3947
Fu, Z., Wang, L., Li, S., Chen, F., Au-Yeung, K. K., & Shi, C. (2021). MicroRNA as an Important Target for Anticancer Drug Development. Frontiers in Pharmacology, 12, 736323. https://doi.org/10.3389/fphar.2021.736323
Hong, D. S., Kang, Y. K., Borad, M., Sachdev, J., Ejadi, S., Lim, H. Y., Brenner, A. J., Park, K., Lee, J. L., Kim, T. Y., Shin, S., Becerra, C. R., Falchook, G., Stoudemire, J., Martin, D., Kelnar, K., Peltier, H., Bonato, V., Bader, A. G., Smith, S., Kim, S., O’Neill, V., & Beg, M. S. (2020, May). Phase 1 study of MRX34, a liposomal miR-34a mimic, in patients with advanced solid tumours. British Journal of Cancer, 122(11), 1630-1637. https://doi.org/10.1038/s41416-020-0802-1
Magen, I., Yacovzada, N. S., Yanowski, E., Coenen-Stass, A., Grosskreutz, J., Lu, C. H., Greensmith, L., Malaspina, A., Fratta, P., & Hornstein, E. (2021, Nov). Circulating miR-181 is a prognostic biomarker for amyotrophic lateral sclerosis. Nature Neuroscience, 24(11), 1534-1541. https://doi.org/10.1038/s41593-021-00936-z
Nemeth, K., Bayraktar, R., Ferracin, M., & Calin, G. A. (2024, Mar). Non-coding RNAs in disease: from mechanisms to therapeutics. Nat Rev Genet, 25(3), 211-232. https://doi.org/10.1038/s41576-023-00662-1
O’Brien, J., Hayder, H., Zayed, Y., & Peng, C. (2018). Overview of MicroRNA Biogenesis, Mechanisms of Actions, and Circulation. Frontiers in Endocrinology, 9, 402. https://doi.org/10.3389/fendo.2018.00402
O’Neill, C. P., & Dwyer, R. M. (2020, Feb 24). Nanoparticle-Based Delivery of Tumor Suppressor microRNA for Cancer Therapy. Cells, 9(2). https://doi.org/10.3390/cells9020521
Seyhan, A. A. (2024, Jan 25). Trials and Tribulations of MicroRNA Therapeutics. Int J Mol Sci, 25(3). https://doi.org/10.3390/ijms25031469
Shah, R. V., Rong, J., Larson, M. G., Yeri, A., Ziegler, O., Tanriverdi, K., Murthy, V., Liu, X., Xiao, C., Pico, A. R., Huan, T., Levy, D., Lewis, G. D., Rosenzweig, A., Vasan, R. S., Das, S., & Freedman, J. E. (2018, Sep 1). Associations of Circulating Extracellular RNAs With Myocardial Remodeling and Heart Failure. JAMA Cardiol, 3(9), 871-876. https://doi.org/10.1001/jamacardio.2018.2371
Xu, W. D., Feng, S. Y., & Huang, A. F. (2022, Dec). Role of miR-155 in inflammatory autoimmune diseases: a comprehensive review. Inflammation Research, 71(12), 1501-1517. https://doi.org/10.1007/s00011-022-01643-6
Foto de portada: Niklas Elmehed / Fundación Nobel