La COVID-19, enfermedad pandémica causada por el nuevo coronavirus SARS CoV-2 (del inglés, virus del síndrome respiratorio agudo severo coronavirus 2) ha causado una crisis económica y social en el mundo. El número de pérdidas humanas y personas con secuelas permanentes, han acelerado la búsqueda de fármacos para su tratamiento, así como el desarrollo de vacunas para el control de la infección. Para este objetivo, el conocimiento y caracterización de los componentes proteicos codificados por su genoma, no sólo de las más inmunogénicas, sino aquellas que induzcan una respuesta inmune protectora, son elementos cruciales para el diseño, desarrollo e implementación de vacunas (Astuti & Ysrafil, 2020).
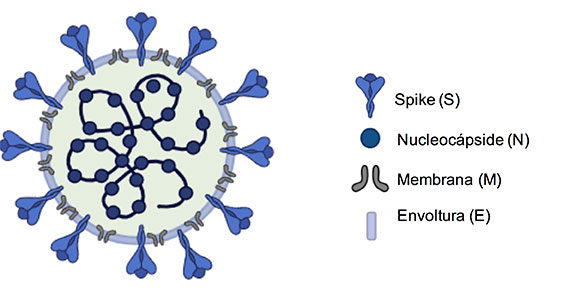
Por ello es importante recordar que el genoma de SARS CoV-2 codifica para cuatro proteínas estructurales: Espiga (S), Membrana (M), Envoltura (E) y Nucleocápside (N) (Figura1). La proteína espiga interviene al inicio de la infección, ya que es la molécula que se une al receptor celular de la enzima convertidora de angiotensina (ACE2) a través de una región conocida como dominio de unión al receptor (RBD). Así RBD es considerado el principal antígeno blanco en el diseño y elaboración de vacunas para SARS CoV-2 (Astuti & Ysrafil, 2020; Zhou et al., 2020).
Existe una gran variedad de estrategias para el desarrollo de vacunas contra SARS CoV-2, tales como vacunas vivas, atenuadas, basadas en DNA o RNA entre otras. Muchos candidatos vacunales aprobados involucran a la proteína de espiga completa o, a una porción de ésta. Hasta el momento se desconoce con suficiente detalle si alguna otra proteína de SARS CoV-2 en combinación con la proteína S, podrían ser utilizadas para dar mejor protección contra el virus en cuestión (Jain et al., 2020).
Estudios en las poblaciones afectadas por las pandemias ocasionadas por otros virus miembros de la misma familia, como el virus del síndrome respiratorio agudo severo o el del síndrome respiratorio de oriente medio (SARS CoV-1 o MERS, por sus siglas en inglés respectivamente), demuestran que la proteína N de estos coronavirus, es una molécula altamente inmunogénica que se expresa con abundancia durante el curso de la infección. Adicional a esto, un estudio reciente demostró que los anticuerpos inducidos por la proteína N son indicadores más sensibles que los anticuerpos anti-espiga para la detección temprana de la infección de SARS CoV-2 (Burbelo et al., 2020).
Algunos grupos de trabajo han evaluado el grado de inmunogenicidad de la proteína N en modelos preclínicos para incluirlo en los candidatos vacunales. Existe evidencia de que una vacuna de DNA que codifica para la proteína N fue capaz de inducir una respuesta humoral y celular robusta en un modelo de ratones C57/BL6 (Zhao et al., 2005).
Una investigación aún más alentadora se basa en un estudio publicado por Hong y colaboradores en el Journal of Science Advances, en donde demostraron que la inmunogenicidad de las proteínas RBD y N, se veía incrementada al evaluar el efecto adyuvante del epítopo P2 del toxoide tetánico (RBD-P2, N-P2) observando un efecto benéfico de la proteína N en la vacuna. El análisis se realizó en tres diferentes especies de animales, ratón, rata y primates no humanos; se observó un incremento en la respuesta inmune celular, mediada por células T y un aumento en la expresión de moléculas de activación, como CD69 en células T CD4+ así como en la producción de citocinas importantes en la inmunidad celular y humoral, como IFNγ e IL-4 respectivamente. Las mismas moléculas RBD-P2 y RBD-P2/N al ser administradas en primates no humanos fueron capaces de protegerlos ante el reto con SARS CoV-2. Además, se encontró un aumento en la producción de IFNγ, así como en el número de células productoras de IFNγ. Sin embargo, los investigadores notaron que la producción de citocinas perfil tipo Th2 (IL-4, IL-5 e Il-13) fue muy baja. De igual manera se observó, en muestras de garganta analizadas en un frotis, que la eliminación del virus fue más rápida cuando se inmunizó con RBD-P2/N. Por lo tanto, el incremento en la activación de células T aunado a una respuesta eficiente de tipo TH1 podría resultar en una eliminación del virus mucho más eficiente, así como en el control de la diseminación viral (Hong et al., 2021).
Por lo tanto, la proteína de nucleocápside (N) ha sido propuesta como un blanco vacunal eficiente, debido a que se observaron células T especificas contra N tanto en ratones inmunizados como en pacientes convalecientes de COVID-19 (Hong et al., 2021).
Finalmente, una ventaja del uso de la proteína N y su inclusión o combinación en los esquemas de inmunización es que la proteína S contiene muchas mutaciones que dan origen a variantes (alrededor de 30 mutaciones), mientras que la secuencia de aminoácidos de la proteína N es más conservada, por lo tanto, podría proporcionar una protección más eficaz contra las variantes de preocupación del SARS CoV-2 (Variantes que posiblemente escapen a la respuesta inmune del organismo, tales como: Delta y Ómicron) (Hong et al., 2021).
En el estudio de Matchett y colaboradores, en 2021, se empleó el vector Adenoviral Ad5 que expresa la proteína N para inmunizar hámsters sirios. Los investigadores observaron la inducción de células T y la supervivencia de estos animales tras someterlos a un reto con la variante Alfa del SARS CoV-2. Por lo anterior, este grupo propone que la proteína N induce una respuesta inmune independiente de la capacidad de neutralización de los anticuerpos inducidos por la proteína S, siendo la respuesta inmune contra la proteína N muy eficiente contra las variantes del SARS CoV-2 (Matchett et al., 2021).
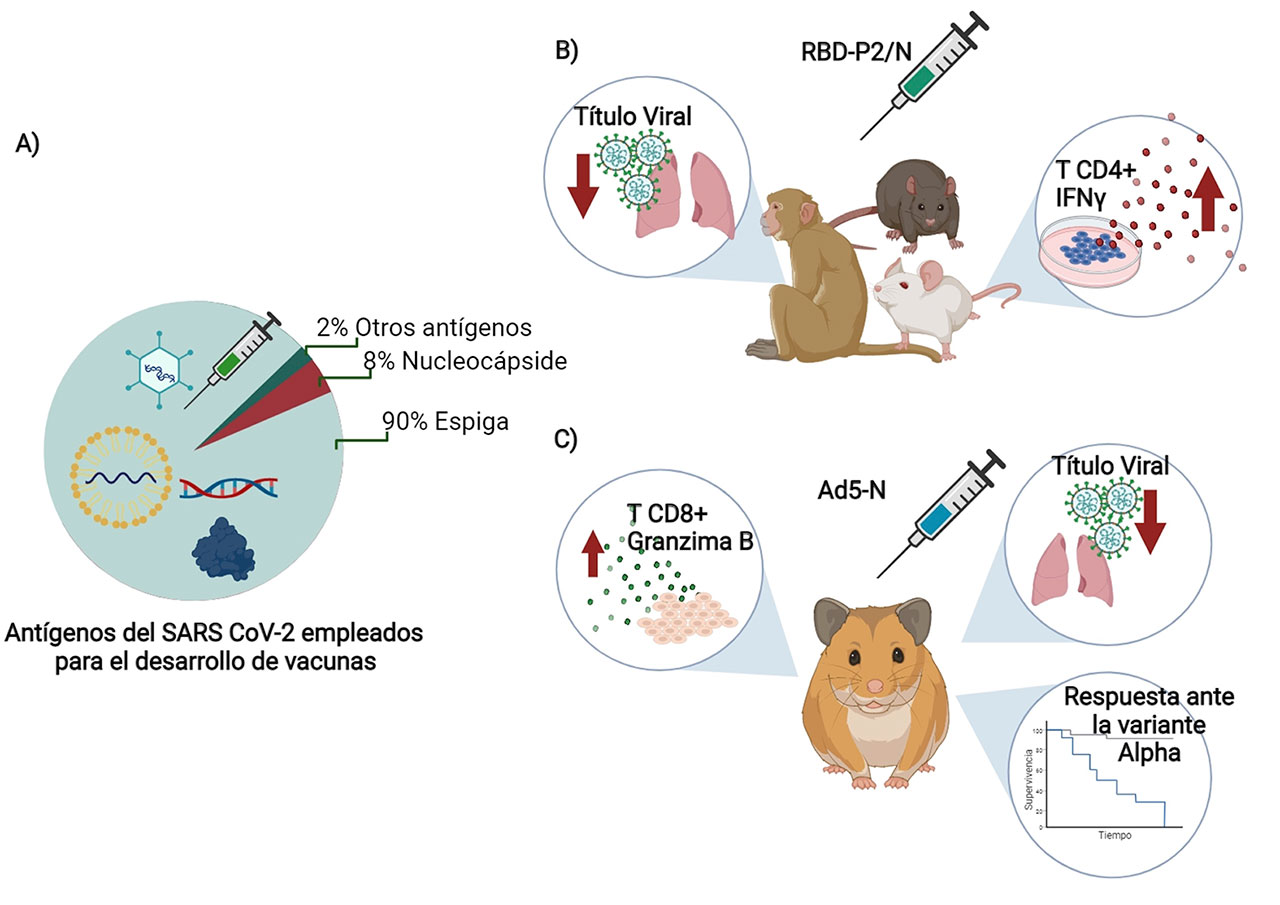
De tal suerte, la combinación de las proteínas RBD/N en la inmunización podría traer una ventaja en la respuesta inmune celular, al superar los mecanismos de escape viral, conferidos por las mutaciones en la proteína S. (Figura 2) (Hong et al., 2021; Matchett et al., 2021).
Referencias
Astuti, I., & Ysrafil. (2020). Severe Acute Respiratory Syndrome Coronavirus 2 (SARS-CoV-2): An overview of viral structure and host response. Diabetes and Metabolic Syndrome: Clinical Research and Reviews, 14(4), 407–412. https://doi.org/10.1016/j.dsx.2020.04.020
Burbelo, P. D., Riedo, F. X., Morishima, C., Rawlings, S., Smith, D., Das, S., Strich, J. R., Chertow, D. S., Davey, R. T., & Cohen, J. I. (2020). Sensitivity in detection of antibodies to nucleocapsid and spike proteins of severe acute respiratory syndrome coronavirus 2 in patients with coronavirus disease 2019. Journal of Infectious Diseases, 222, 206–213. https://doi.org/10.1093/infdis/jiaa273
Hong, S. H., Oh, H., Park, Y. W., Kwak, H. W., Oh, E. Y., Park, H. J., Kang, K. W., Kim, G., Koo, B. S., Hwang, E. H., Baek, S. H., Park, H. J., Lee, Y. S., Bang, Y. J., Kim, J. Y., Bae, S. H., Lee, S. J., Seo, K. W., Kim, H., … Nam, J. H. (2021). Immunization with RBD-P2 and N protects against SARS-CoV-2 in nonhuman primates. Science Advances, 7(22). https://doi.org/10.1126/SCIADV.ABG7156
Jain, S., Batra, H., Yadav, P., & Chand, S. (2020). COVID-19 Vaccines Currently under Preclinical and Clinical Studies, and Associated Antiviral Immune Response. Vaccines, 8(4), 1–16. https://doi.org/10.3390/VACCINES8040649
Matchett, W. E., Joag, V., Stolley, J. M., Shepherd, F. K., Quarnstrom, C. F., Mickelson, C. K., Wijeyesinghe, S., Soerens, A. G., Becker, S., Thiede, J. M., Weyu, E., O’Flanagan, S., Walter, J. A., Vu, M. N., Menachery, V. D., Bold, T. D., Vezys, V., Jenkins, M. K., Langlois, R. A., & Masopust, D. (2021). Nucleocapsid vaccine elicits spike-independent SARS-CoV-2 protective immunity. BioRxiv : The Preprint Server for Biology. https://doi.org/10.1101/2021.04.26.441518
Zhao, P., Cao, J., Zhao, L. J., Qin, Z. L., Ke, J. S., Pan, W., Ren, H., Yu, J. G., & Qi, Z. T. (2005). Immune responses against SARS-coronavirus nucleocapsid protein induced by DNA vaccine. Virology, 331(1), 128–135. https://doi.org/10.1016/j.virol.2004.10.016
Zhou, P., Yang, X. Lou, Wang, X. G., Hu, B., Zhang, L., Zhang, W., Si, H. R., Zhu, Y., Li, B., Huang, C. L., Chen, H. D., Chen, J., Luo, Y., Guo, H., Jiang, R. Di, Liu, M. Q., Chen, Y., Shen, X. R., Wang, X., … Shi, Z. L. (2020). A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature, 579(7798), 270–273. https://doi.org/10.1038/S41586-020-2012-7