El Adenocarcinoma Ductal Pancreático (PDAC, por sus siglas en inglés), es uno de los cánceres más letales. En México, el PDAC ocupa el 8vo lugar en mortalidad con un índice de fatalidad del 0.96%, es decir, el 96% de los pacientes mueren en un periodo menor a 6 meses (Sung et al., 2021). Esto se debe principalmente a que los pacientes son diagnosticados en etapas avanzadas o metastásicas (80% de los casos), cuando ya no son elegibles para cirugía (Sung et al., 2021). Ya que las terapias comúnmente empleadas contra esta enfermedad [i.e. la gemcitabina (GEM), 5 Fluorouracil] son poco efectivas, el mejoramiento en el tratamiento de este cáncer es un objetivo médico urgente. La mayoría de los esfuerzos de investigación se han centrado en dilucidar cómo las alteraciones genéticas contribuyen al desarrollo del PDAC, con la idea de identificar moléculas clave que puedan ser empleadas para su tratamiento.
En la actualidad, se sabe que la expresión alterada de genes en células cancerosas no solamente es mediada por cambios genéticos sino también por mecanismos epigéneticos, que se han atribuido a alteraciones en los patrones de metilación del ADN y más recientemente en las histonas. Éstas surgen como reguladores críticos (Vardabasso et al., 2014) ya que pueden alterar el paisaje epigenómico mediante dos mecanismos: (1) modificaciones postraduccionales (PTM), como metilación, acetilación, ubiquitinación y fosforilación; y (2) substitución por variantes. La incorporación de éstas en los nucleosomas es uno de los mecanismos epigenéticos más importantes y responsables de modificar la estructura local de la cromatina y de regular procesos celulares como la expresión de genes (Colino-Sanguino et al., 2022; Vardabasso et al., 2014). Su desregulación puede contribuir al inicio y a la progresión del cáncer. (Vardabasso et al., 2014). Por lo tanto, estudiar la expresión y las funciones de las variantes de histonas contribuirá a una mejor comprensión de los mecanismos que favorecen el desarrollo de PDAC.
¿Cómo ocurre esto? Primero hay que recordar que el ADN, en células eucariontes, se encuentra dentro del núcleo asociado a un complejo de proteínas conocidas como histonas canónicas (dos pares de H2A, H2B, H3 Y H4), que favorecen el empaquetamiento del ADN dentro del espacio nuclear. Estas histonas canónicas pueden ser reemplazadas por variantes, las cuales alteran la estructura, estabilidad y dinámica del nucleosoma, afectando así la accesibilidad al ADN y su reparación, y la formación de hetero y eucromatina (Weber & Henikoff, 2014). La incorporación de las variantes de histonas al nucleosoma puede generar estados regulatorios diferentes, lo cual crea un estado cromatínico único que conlleva a controlar funciones específicas dependientes de cada variante.(Weber & Henikoff, 2014).
Una de las variantes de histonas más estudiada y que se halla en diferentes organismos es H2A.Z, la cual presenta un 60% de identidad con su histona canónica H2A (Colino-Sanguino et al., 2022). Se han identificado tres isoformas de H2A.Z, de las cuales dos son transcritas por los genes no alélicos H2AFZ y H2AFV (llamados H2A.Z.1 y H2A.Z.2, respectivamente); y la tercera, denominada H2A.Z.2.2, que resulta del “splicing” alternativo de H2A.Z.2. Ninguna de estas isoformas tienen funciones redundantes (Dryhurst et al., 2009). H2A.Z se incorpora a los nucleosomas y se distribuye en los elementos reguladores del ADN, como promotores y potenciadores (Surface et al., 2016; Valdes-Mora et al., 2017); transcripción de genes activos e inactivos, renovación de nucleosomas, reparación de ADN, límites de heterocromatina, segregación cromosómica, recombinación meiótica, progresión a través del ciclo celular y diferenciación de células madre embrionarias (ESC) (Colino-Sanguino et al., 2022).
Además, se ha demostrado que el agotamiento de H2A.Z en líneas celulares de fibroblastos induce un fenotipo de senescencia al permitir la sobreexpresión de p21 dependiente de p53 (Gevry et al., 2007). Sin embargo, aún se desconoce el papel de H2A.Z en la generación de un transcriptoma asociado con la senescencia en el cáncer. Dada la amplia variedad de funciones que realiza H2A.Z y la complejidad resultante de la presencia de tres isoformas diferentes, no es extraño que la sobreexpresión de H2A.Z sea crucial para el desarrollo de varios tipos de cáncer (Colino-Sanguino et al., 2022), incluido el cáncer de mama (Svotelis et al., 2010), el cáncer de vejiga (Kim et al., 2013) colangicarcinoma intrahepático (ICC) (Yang et al., 2018) y más recientemente el melanoma (cáncer de piel) (Vardabasso et al., 2016). Por ello decidimos estudiar la participación de la histona H2A.Z en el desarrollo del PDAC. Inicialmente se procedió a determinar si la histona H2A.Z se encuentra incrementada en tejido pancreático de pacientes con PDAC, así como en tres líneas celulares de PDAC (PANC-1, Capan-1, y MiaPaCa-2).
Nuestros datos demostraron que H2A.Z está aumentada en líneas celulares de cáncer pancreático, así como en biopsias de pacientes con PDAC. y que sus altos niveles de expresión correlacionan con la progresión de la enfermedad y un mal pronóstico de la misma. Ensayos de RT-qPCR en líneas celulares de PDAC y en tejidos de pacientes con este cáncer demostraron que las tres isoformas de H2AZ a nivel de transcrito están incrementadas. Además de que el “knock down” de las tres isoformas de H2A.Z (3KDH2A.Z) en la línea celular de cáncer pancreático PANC-1, induce un fenotipo senescente, que conlleva a que las células se arresten en la fase G2/M, aumenten la expresión del inhibidor de la cinasa dependiente de ciclina CDKN2A/p16, incrementen su actividad de SA-β-galactosidasa y la producción de la interleucina 8. Asimismo, el análisis del transcriptoma de las células 3KDH2AZ, mostró una expresión génica alterada en las vías de biosíntesis de ácidos grasos y en las que regulan el ciclo celular, así como la reparación del daño en el ADN. Todos, mecanismos implicados en senescencia (Zhou et al., 2021). Es importante destacar que el 3KDH2A.Z, reduce el tamaño del tumor en un modelo de xenoinjerto de ratón in vivo y sensibiliza a las células PDAC a la GEM. También se encontró que el incremento de las isoformas H2A.Z.1 y H2A.Z.2.1 más que el de H2A.Z.2.2, restaura parcialmente el fenotipo oncogénico (Avila-Lopez et al., 2021).
En conclusión, nuestros datos sugieren que la sobreexpresión de las tres isoformas de H2A.Z permiten a las células superar la barrera oncoprotectora asociada con la senescencia, favoreciendo el crecimiento del tumor PDAC y la quimiorresistencia. Así, estos resultados convierten a H2A.Z en un regulador epigenético clave en la biología del PDAC, y en una mólecula promisoria para el pronóstico y blanco terapéutico contra este tipo de cáncer (Figura 1).
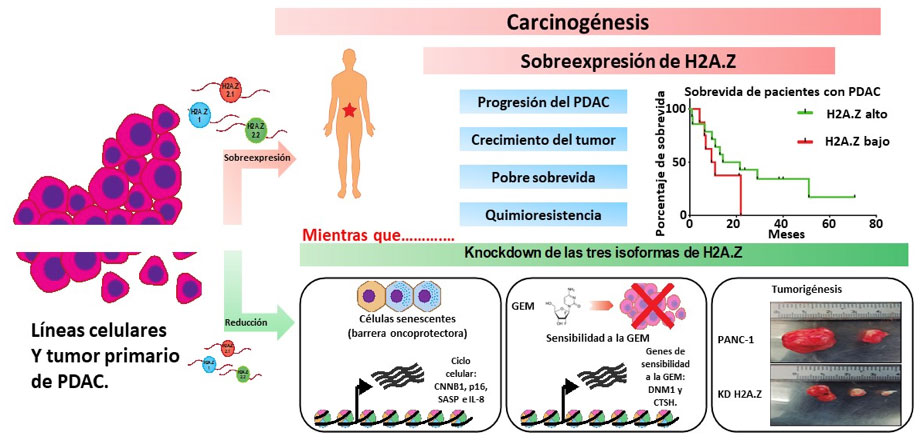
En pacientes con PDAC, las tres isoformas de H2A.Z se encuentran incrementadas, lo que se se asocia con la progresión, el crecimiento del tumor y con una pobre sobrevida de los pacientes, por lo que H2A.Z resulta ser un potencial biomarcador de diagnóstico y mal pronóstico de la enfermedad (imágen superior). Asimismo, H2A.Z podría ser un blanco terapéutico prometedor, pues la reducción de los niveles de expresión de sus tres isoformas “knock down” reduce el fenotipo proliferativo de las células cancerosas, aumenta la sensibilidad a la GEM y reduce el tamaño del tumor (imágen inferior).
Referencias
Avila-Lopez, P. A., Guerrero, G., Nunez-Martinez, H. N., Peralta-Alvarez, C. A., Hernandez-Montes, G., Alvarez-Hilario, L. G., Herrera-Goepfert, R., Albores-Saavedra, J., Villegas-Sepulveda, N., Cedillo-Barron, L., Montes-Gomez, A. E., Vargas, M., Schnoor, M., Recillas-Targa, F., & Hernandez-Rivas, R. (2021). H2A.Z overexpression suppresses senescence and chemosensitivity in pancreatic ductal adenocarcinoma. Oncogene, 40(11), 2065-2080. https://doi.org/10.1038/s41388-021-01664-1
Colino-Sanguino, Y., Clark, S. J., & Valdes-Mora, F. (2022). The H2A.Z-nucleosome code in mammals: emerging functions. Trends Genet, 38(5), 516. https://doi.org/10.1016/j.tig.2022.02.004
Dryhurst, D., Ishibashi, T., Rose, K. L., Eirin-Lopez, J. M., McDonald, D., Silva-Moreno, B., Veldhoen, N., Helbing, C. C., Hendzel, M. J., Shabanowitz, J., Hunt, D. F., & Ausio, J. (2009). Characterization of the histone H2A.Z-1 and H2A.Z-2 isoforms in vertebrates. BMC Biol, 7, 86. https://doi.org/10.1186/1741-7007-7-86
Gevry, N., Chan, H. M., Laflamme, L., Livingston, D. M., & Gaudreau, L. (2007). p21 transcription is regulated by differential localization of histone H2A.Z. Genes Dev, 21(15), 1869-1881. https://doi.org/10.1101/gad.1545707
Kim, K., Punj, V., Choi, J., Heo, K., Kim, J. M., Laird, P. W., & An, W. (2013). Gene dysregulation by histone variant H2A.Z in bladder cancer. Epigenetics Chromatin, 6(1), 34. https://doi.org/10.1186/1756-8935-6-34
Sung, H., Ferlay, J., Siegel, R. L., Laversanne, M., Soerjomataram, I., Jemal, A., & Bray, F. (2021). Global Cancer Statistics 2020: GLOBOCAN Estimates of Incidence and Mortality Worldwide for 36 Cancers in 185 Countries. CA Cancer J Clin, 71(3), 209-249. https://doi.org/10.3322/caac.21660
Surface, L. E., Fields, P. A., Subramanian, V., Behmer, R., Udeshi, N., Peach, S. E., Carr, S. A., Jaffe, J. D., & Boyer, L. A. (2016). H2A.Z.1 Monoubiquitylation Antagonizes BRD2 to Maintain Poised Chromatin in ESCs. Cell Rep, 14(5), 1142-1155. https://doi.org/10.1016/j.celrep.2015.12.100
Svotelis, A., Gevry, N., Grondin, G., & Gaudreau, L. (2010). H2A.Z overexpression promotes cellular proliferation of breast cancer cells. Cell Cycle, 9(2), 364-370. https://doi.org/10.4161/cc.9.2.10465
Valdes-Mora, F., Gould, C. M., Colino-Sanguino, Y., Qu, W., Song, J. Z., Taylor, K. M., Buske, F. A., Statham, A. L., Nair, S. S., Armstrong, N. J., Kench, J. G., Lee, K. M. L., Horvath, L. G., Qiu, M., Ilinykh, A., Yeo-Teh, N. S., Gallego-Ortega, D., Stirzaker, C., & Clark, S. J. (2017). Acetylated histone variant H2A.Z is involved in the activation of neo-enhancers in prostate cancer. Nat Commun, 8(1), 1346. https://doi.org/10.1038/s41467-017-01393-8
Vardabasso, C., Hake, S. B., & Bernstein, E. (2016). Histone variant H2A.Z.2: A novel driver of melanoma progression. Mol Cell Oncol, 3(2), e1073417. https://doi.org/10.1080/23723556.2015.1073417
Vardabasso, C., Hasson, D., Ratnakumar, K., Chung, C. Y., Duarte, L. F., & Bernstein, E. (2014). Histone variants: emerging players in cancer biology. Cell Mol Life Sci, 71(3), 379-404. https://doi.org/10.1007/s00018-013-1343-z
Weber, C. M., & Henikoff, S. (2014). Histone variants: dynamic punctuation in transcription. Genes Dev, 28(7), 672-682. https://doi.org/10.1101/gad.238873.114
Yang, B., Tong, R., Liu, H., Wu, J., Chen, D., Xue, Z., Ding, C., Zhou, L., Xie, H., Wu, J., & Zheng, S. (2018). H2A.Z regulates tumorigenesis, metastasis and sensitivity to cisplatin in intrahepatic cholangiocarcinoma. Int J Oncol, 52(4), 1235-1245. https://doi.org/10.3892/ijo.2018.4292
Zhou, D., Borsa, M., & Simon, A. K. (2021). Hallmarks and detection techniques of cellular senescence and cellular ageing in immune cells. Aging Cell, 20(2), e13316. https://doi.org/10.1111/acel.13316













1 comentario
Good Job!
Comentarios no permitidos.