A medida que avanzan la ciencia y la tecnología se ha comenzado a buscar distintas alternativas para la fabricación, creación y desarrollo de nuevos fármacos que coadyuven en el tratamiento de diferentes enfermedades. Dentro de estas opciones se encuentra la reutilización o reposicionamiento de fármacos ya existentes. Ante la reciente pandemia de COVID-19, lo anterior ha cobrado gran relevancia para el tratamiento de ésta y otras enfermedades.
La COVID-19 ha causado gran conmoción en la sociedad, ya que de acuerdo con las cifras de la Organización Mundial de la Salud (OMS), se reportaron casi 3 millones de muertes en el continente americano entre 2020 y 2023.1 Ante el padecimiento de COVID-19 no se ha logrado establecer un tratamiento específico que ataque directamente al virus SARS-CoV-2; por ende, surge la necesidad de desarrollar tratamientos dirigidos contra este virus.2 Sin embargo, el desarrollo de medicamentos específicos que actúen contra el virus, así como la realización de investigaciones de laboratorio en modelos experimentales y animales para evaluar su eficacia, implican procesos prolongados y costosos. Por tanto, no constituyen una estrategia viable para proporcionar tratamientos a corto plazo con el fin de contener una pandemia de gran magnitud e impacto en términos de morbilidad y mortalidad.
Además, tras superar la fase de estudios y pruebas de laboratorio, es necesario aplicar estos medicamentos experimentales en pacientes, así como en entornos clínicos que incluyen salas de hospitalización, médicos y personal de enfermería, para proceder con la fase clínica del proceso experimental. Este paso es crucial para evaluar tanto la efectividad del tratamiento contra la enfermedad como su seguridad para los pacientes. Este enfoque se traduce en un alto costo socioeconómico, lo que a menudo impide que sea considerado una alternativa terapéutica rápida y eficaz ante una pandemia que avanza rápidamente. Es aquí donde nace la idea de reutilizar fármacos ya existentes, usados para el tratamiento de otras patologías, pero que han demostrado tener un efecto contra SARS-CoV-2.
Dos de las metodologías en experimentación in silico (es decir, a través de la simulación computacional), para la reutilización de fármacos, son el docking molecular y el virtual screening. Éstas han causado una gran expectativa en diferentes campos de las ciencias aplicadas, pues han destacado como herramientas valiosas en la búsqueda de medicamentos para el tratamiento de la enfermedad. El docking molecular es una técnica de modelado computacional que permite predecir la interacción entre moléculas, como fármacos y proteínas virales, lo que lo convierte en un enfoque prometedor para la reutilización de fármacos existentes en la lucha contra COVID-19.3
El docking molecular se basa en la simulación donde se correlaciona la unión de una molécula pequeña, ya sea un fármaco, con una macromolécula como una proteína: en este caso, una proteína viral. Lo que se pretende con esta unión es impedir la entrada del virus o la replicación en el huésped. Utiliza métodos de cálculo para predecir las interacciones no covalentes entre las moléculas, como enlaces de hidrógeno, interacciones iónicas y fuerzas de Van der Waals.4-5 El objetivo principal del docking es predecir la geometría, topología y la energía de unión más probable entre el fármaco y la proteína, lo que puede conducir a la identificación de candidatos a medicamentos potenciales.
Este proceso es como un juego de encaje, donde se intenta ver cómo una molécula pequeña, que podría ser un medicamento, se ajusta dentro de una parte específica de una proteína relacionada con una enfermedad. Imagina que la proteína es como una cerradura y el medicamento potencial es como una llave; solo las llaves que encajan perfectamente pueden abrir la cerradura y, en este caso, combatir la enfermedad.
La “energía de unión” se refiere a qué tan fuerte es la conexión entre la llave (el medicamento potencial) y la cerradura (la proteína). Si la energía de unión es alta, significa que la llave encaja muy bien y es probable que permanezca en su lugar, actuando eficazmente. Por lo tanto, una molécula con una alta energía de unión es considerada un buen candidato para ser un medicamento porque indica que puede unirse firmemente a la proteína objetivo, bloqueando o modificando su función de manera eficaz para tratar la enfermedad.
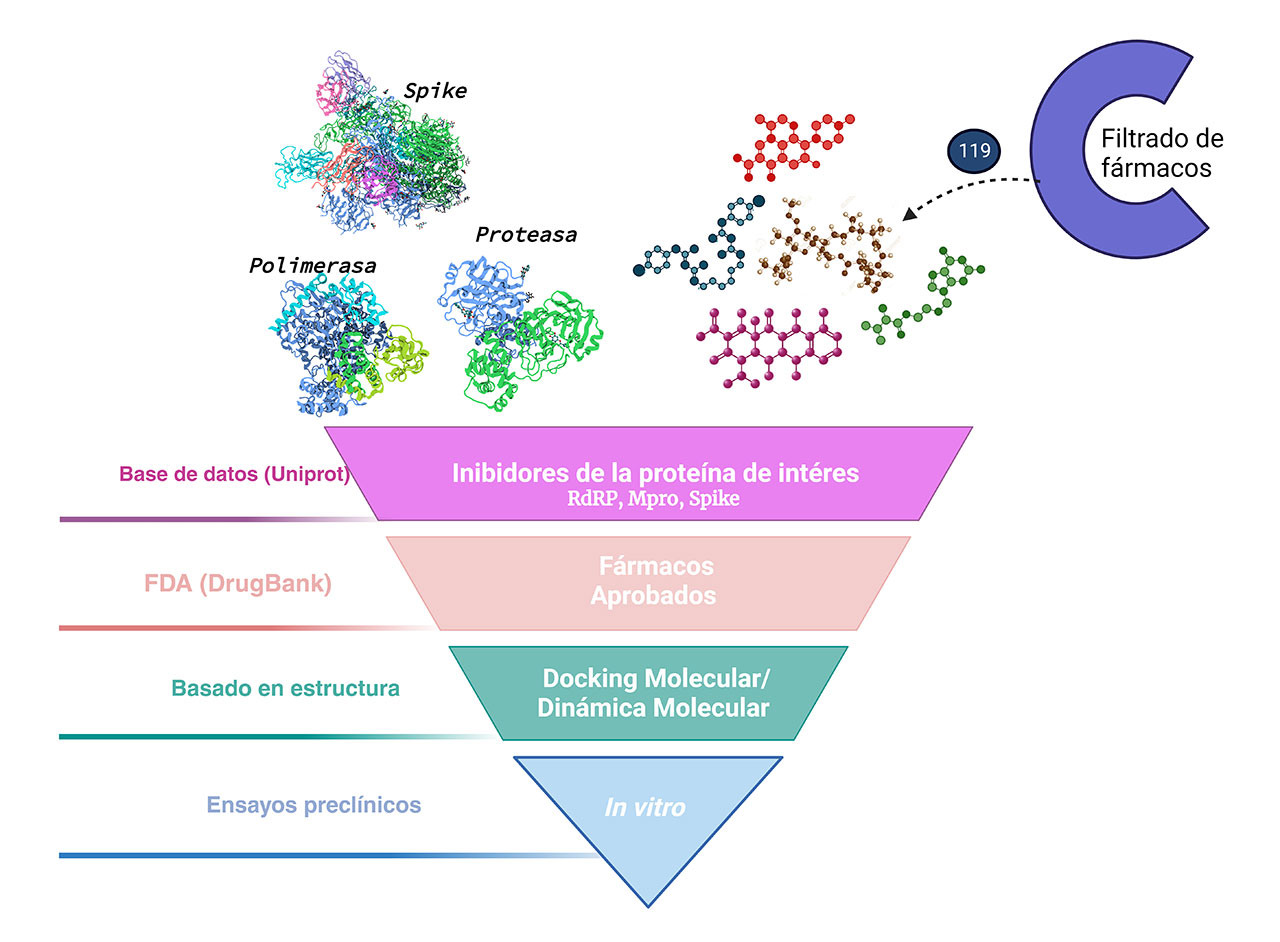
Por otro lado, el virtual screening se define como un método que a través de una colección de fármacos y el uso del docking molecular, identifica cuáles son los fármacos más probables para unirse a un objetivo como las proteínas virales. Esta metodología utiliza algoritmos matemáticos para examinar un gran número de moléculas con capacidad de interactuar con una diana de interés específico (Figura 1).
Estas metodologías empleadas en la reutilización de fármacos permiten evaluar la capacidad de fármacos existentes, que ya han sido aprobados para otras enfermedades desde un sistema de cómputo con características específicas. Su desventaja es que son simulaciones computacionales que necesitan ser probadas experimentalmente.
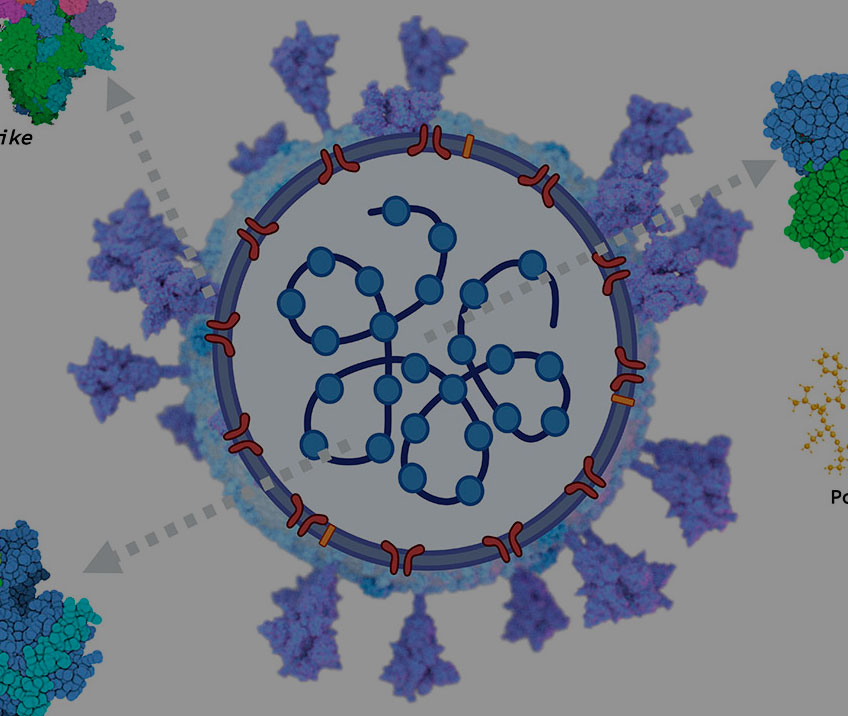
Particularmente se han identificado tres blancos farmacológicos importantes en el virus SARS-CoV-2, como la proteína spike que interviene en la unión del virus a la célula huésped; el segundo es la proteasa principal (Mpro o NSP5) encargada de procesar la poliproteína del virus generando proteínas únicas funcionales; y finalmente, la RNA-polimerasa dependiente de RNA (RdRp NSP12) que se ocupa de la replicación del genoma viral.6
Teniendo en cuenta estos tres blancos farmacológicos, y las metodologías del virtual screening, recientemente se han llevado a cabo diversos estudios enfocados en la reutilización de fármacos contra el virus de SARS-CoV-2. Brevemente, realizamos una búsqueda científica usando el motor PubMed, encontrando 119 estudios orientados a la reutilización de fármacos con actividad anti-SARS-CoV-2, que usan el virtual screening y el docking molecular para este fin.
Entre los artículos de reutilización de fármacos como tratamiento para la COVID-19 (Figura 2), se encuentra el de Mohammad A Elmorsy y colaboradores en 2021, quienes llevaron a cabo una metodología de experimentación in silico con el programa Molecular Operating Environment (MOE). En este modelo se seleccionaron diferentes fármacos aprobados por la administración de alimentos y medicamentos de Estados Unidos (FDA), dirigidos contra diversas proteínas del virus SARS-CoV-2. De acuerdo con este modelo in silico, uno de los mejores resultados obtenidos se refiere a la polimixina B, dirigida contra la proteína spike del virus.7 Este antibiótico es utilizado actualmente contra la bacteria Pseudomonas aeruginosa, causante de la infección del tracto urinario; el medicamento va dirigido contra la membrana de las bacterias gram negativas.8
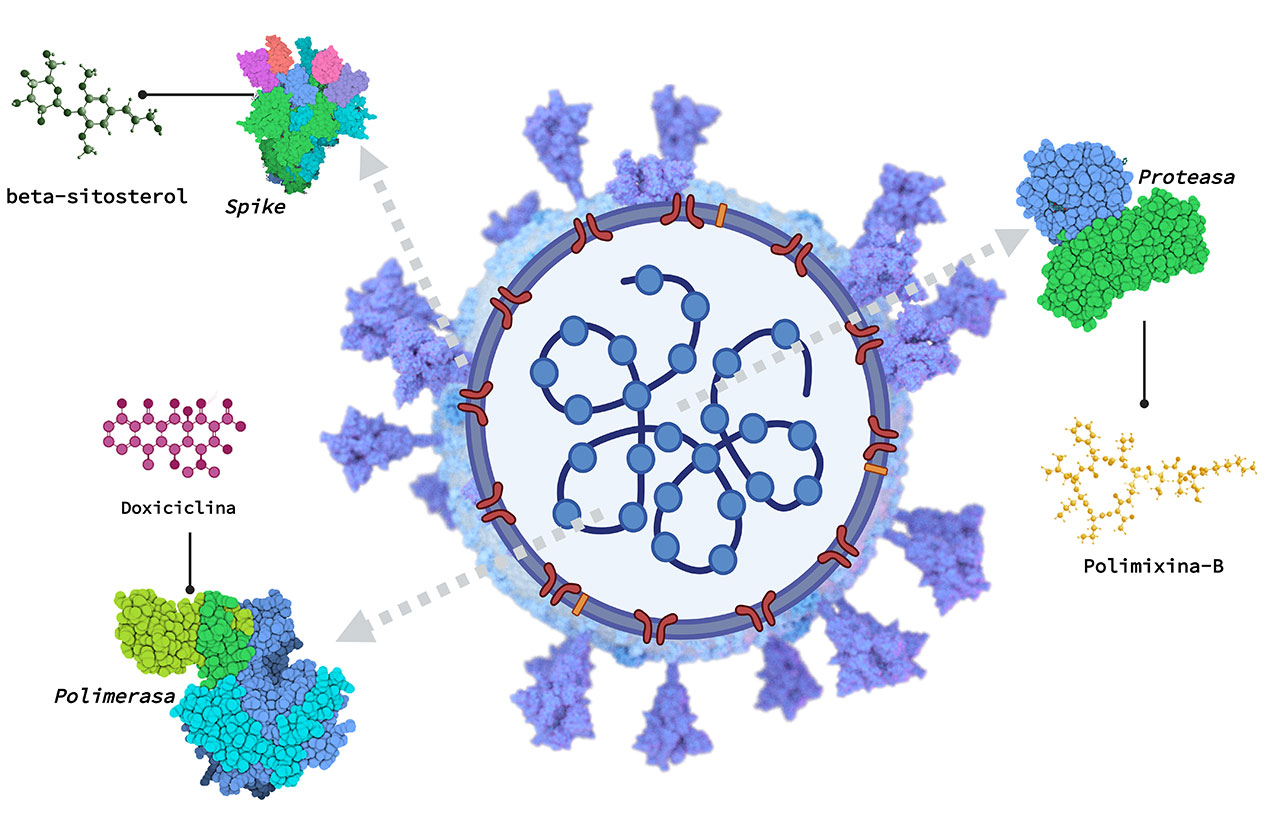
En otro estudio se han realizado análisis in silico e in vitro por parte de Rajaiah Alexpandi en 2022. En esta investigación utilizaron doxiciclina con el fin de disminuir y detener la replicación viral; el enfoque principal fue inhibir la acción de la proteína RNA polimerasa, encargada de la replicación del genoma viral. La doxiciclina demostró tener actividad antiviral contra aislados clínicos de pacientes hospitalizados; este análisis fue realizado durante marzo del 2020, una fecha importante durante el brote pandémico.9
La doxiciclina está indicada como tratamiento de elección en diversas infecciones provocadas por bacterias, transmisión sexual, enfermedad de Lyme e infecciones oftálmicas, debido a que su mecanismo de acción se basa en la inhibición de la síntesis de proteínas, lo cual produce una inhibición en la replicación de los agentes infecciosos.10
Además de utilizar medicamentos aprobados contra otras enfermedades, el virtual screening es una técnica que también se puede usar para estudiar fármacos de creación reciente, como nuevos tratamientos para combatir el virus de SARS-COV2. Gracias a esta metodología se han llevado a cabo distintas investigaciones de diversos grupos para la creación de fármacos a partir de derivados de plantas.
Por ejemplo, en 2021 se llevaron a cabo análisis de diversos fitocompuestos (derivados de plantas), por medio de técnicas de biología molecular. El estudio arrojó resultados como el beta-sitosterol, que pertenece al grupo de esteroides; el fitocompuesto mostró propiedades antivirales contra SARS-CoV-2. Este complejo se encuentra en diversas plantas herbáceas como Curcuma longa, Piper nigrum, y Zingiber officinale, popularmente conocidas como curcuma, pimienta negra y jengibre respectivamente.11 Sin embargo, aun después de estos resultados, se requiere el diseño de estudios in vitro e in vivo para que pueda ser un probado en humanos, y posteriormente, presentado como un fármaco contra la COVID-19.
En conclusión, el virtual screening y el docking molecular son técnicas esenciales que sirven como análisis preliminares en la reutilización de fármacos. Estas metodologías posibilitan la evaluación ágil de compuestos mediante simulaciones computacionales, previas a las fases de estudio in vitro e in vivo.
Esta innovación tecnológica fue especialmente valiosa durante la pandemia de COVID-19, en un contexto donde muchos laboratorios clínicos y de investigaciones especializadas cerraron sus puertas. El estudio computacional del virus permitió profundizar en la identificación de nuevos blancos farmacológicos y sugerir tratamientos alternativos para combatir el SARS-COV2.
Uno de los beneficios más significativos de estas metodologías es la eficiencia en la prueba de fármacos ya aprobados por organismos como la FDA (USA) o la Cofepris en México. Al usar productos previamente validados, se simplifica el proceso, se ahorra tiempo y recursos, facilitando un acceso más rápido a ensayos clínicos.
Es crucial entender que, independientemente de estas técnicas, los ensayos clínicos son indispensables para confirmar la eficiencia y eficacia de cualquier tratamiento propuesto. Aunque la inteligencia artificial ofrece herramientas avanzadas de predicción, simulación y ensayo, no pueden sustituir a las pruebas clínicas o in vivo. Estas tecnologías son excelentes para explorar posibles interacciones con la molécula objetivo, en este caso, contra el virus SARS-COV-2. La validación final de la seguridad y eficacia del fármaco siempre requerirá ensayos experimentales.
Referencias
- World Health Organization.(s.f.).WHO Coronavirus (COVID-19) Dashboard. https://covid19.who.int/
- Jackson CB, Farzan M, Chen B, Choe H.(2022). Mechanisms of SARS-CoV-2 entry into cells. Nat Rev Mol Cell Biol.23(1):3-20.
- Rohban MH, Fuller AM, Tan C, Goldstein JT, Syangtan D, Gutnick A, DeVine A, Nijsure MP, Rigby M, Sacher JR, Corsello SM, Peppler GB, Bogaczynska M, Boghossian A, Ciotti GE, Hands AT, Mekareeya A, Doan M, Gale JP, Derynck R, Turbyville T, Boerckel JD, Singh S, Kiessling LL, Schwarz TL, Varelas X, Wagner FF, Kafri R, Eisinger-Mathason TSK, Carpenter AE.(2022). Virtual screening for small-molecule pathway regulators by image-profile matching. Cell Syst. 21;13(9):724-736.
- Elmorsy MA, El-Baz AM, Mohamed NH, Almeer R, Abdel-Daim MM, Yahya G.(2022). In silico screening of potent inhibitors against COVID-19 key targets from a library of FDA-approved drugs. Environ Sci Pollut Res Int.;29(8):12336-12346.
- Gupta R, Srivastava D, Sahu M, Tiwari S, Ambasta RK, Kumar P.(2021) Artificial intelligence to deep learning: machine intelligence approach for drug discovery. Mol Divers.(3):1315-1360.
- Malone B, Urakova N, Snijder EJ, Campbell EA. Structures and functions of coronavirus replication-transcription complexes and their relevance for SARS-CoV-2 drug design. (2022).Nat Rev Mol Cell Biol.23(1):21-39.
- Elmorsy MA, El-Baz AM, Mohamed NH, Almeer R, Abdel-Daim MM, Yahya G. In silico screening of potent inhibitors against COVID-19 key targets from a library of FDA-approved drugs.(2022). Environ Sci Pollut Res Int. 29(8):12336-12346.
- Rigatto MH, Falci DR, Zavascki AP. (2019).Clinical Use of Polymyxin B. Adv Exp Med Biol. 1145:197-218.
- Alexpandi R, Gendrot M, Abirami G, Delandre O, Fonta I, Mosnier J, Mariadasse R, Jeyakanthan J, Pandian SK, Pradines B, Ravi AV. (2022).Repurposing of Doxycycline to Hinder the Viral Replication of SARS-CoV-2: From in silico to in vitro Validation. Front Microbiol. 4;13:757418.
- Patel RS, Parmar M.(2023). Doxycycline Hyclate. . In: StatPearls. Treasure Island (FL): StatPearls Publishing.
- Sankar M, Ramachandran B, Pandi B, Mutharasappan N, Ramasamy V, Prabu PG, Shanmugaraj G, Wang Y, Muniyandai B, Rathinasamy S, Chandrasekaran B, Bayan MF, Jeyaraman J, Halliah GP, Ebenezer SK.(2021). In silico Screening of Natural Phytocompounds Towards Identification of Potential Lead Compounds to Treat COVID-19. Front Mol Biosci. 5;8:637122.