Foto de Kbradnam de Wikipedia en inglés.
El DNA, contenido en las células, almacena la información requerida para formar a cualquier ser vivo. Esto se realiza mediante la copia de la información codificada en el DNA en RNA (en un proceso llamado transcripción), el cual funciona como molde para sintetizar las proteínas necesarias para que los organismos se desarrollen. Este proceso, por el cual la información codificada en un gen es usada para la formación de proteínas y otros elementos no codificantes, se denomina expresión genética y es altamente regulado dependiendo de las condiciones medioambientales, así como por factores internos. Uno de los elementos endógenos que regulan la expresión genética a nivel post-transcripcional (regulación a nivel de RNA) son los microRNAs (miRNAs).
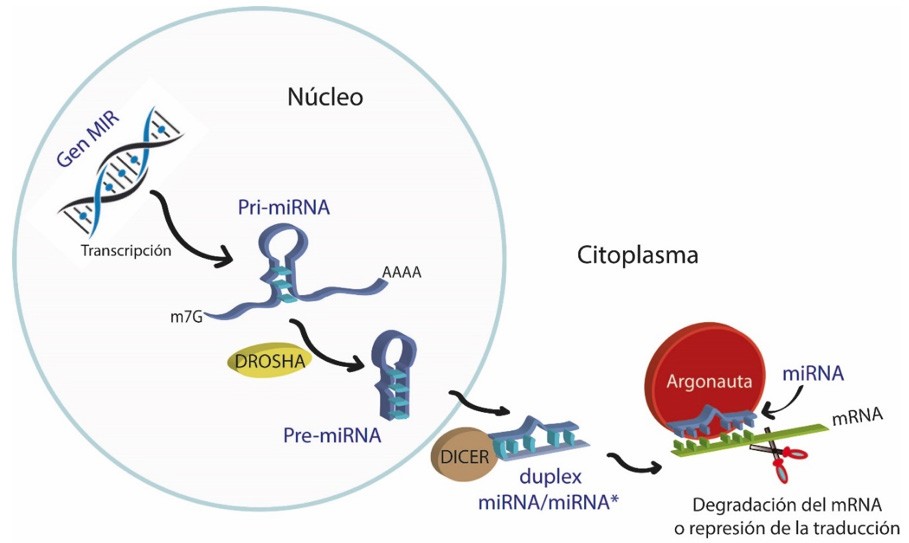
Los miRNAs son pequeñas moléculas de RNA no codificante de apenas 20 a 24 nucleótidos que funcionan como guías para llevar a cabo el silenciamiento génico, es decir reducir o eliminar la expresión de un gen. Los miRNAs pertenecen a una clase de RNAs pequeños (del inglés small RNAs, sRNAs) que derivan de precursores con una estructura tipo tallo-asa que son codificados en el genoma y procesados por proteínas llamadas DROSHA y DICER que reducen su longitud y, así, llevan a cabo su función. La forma en la que actúan los miRNAs es a través de complementariedad con un RNA mensajero (mRNA) y, mediante la asociación con enzimas llamadas ARGONAUTA, impide que este transcrito pueda traducirse en una proteína, ya sea por represión de su traducción o mediante su degradación (Ha and Kim, 2014) (Fig.1).
A pesar de su tamaño, los miRNAs son reguladores maestros de la expresión génica en la mayoría de los organismos eucariotas y están involucrados en diversos procesos desde el desarrollo y crecimiento, hasta su participación en respuesta a estrés o en el desarrollo de enfermedades como el cáncer. En la actualidad existe una basta colección de artículos que describen su función y regulación, por tanto, el objetivo de este escrito es hacer una recapitulación de cómo fue su descubrimiento hace ya más de 20 años y describir sus principales funciones en animales y plantas, así como su aplicación en biomedicina y biotecnología vegetal.
El descubrimiento de los miRNAs: resultado de un modelo biológico perfecto
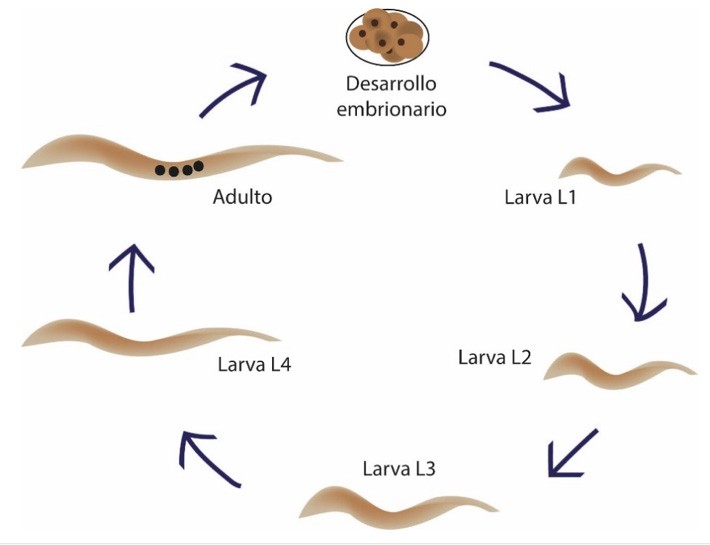
Aunque el descubrimiento del primer miRNA se describe como un evento producto de la suerte y casualidad, en realidad este hallazgo fue el resultado del conocimiento exhaustivo que se tenía del modelo de estudio: el nemátodo Caenorhabditis elegans. Este organismo había sido empleado desde hace mucho tiempo para describir con precisión los mecanismos de control de su desarrollo (Lee et al., 2004). Se sabe que el ciclo de vida de C. elegans incluye una etapa de desarrollo embrionario seguido de cuatro estadios larvales (denominados: L1, L2, L3 y L4) y finalmente su estadio de adulto. Todo su ciclo lo finaliza en aproximadamente tres días, lo que lo vuelve un excelente modelo de estudio (www.wormatlas.org) (Fig.2).
Los investigadores Robert Horvitz del Instituto Tecnológico de Massachussetts (MIT) y John Sulston de la Universidad de Cambridge fueron los encargados de trazar el desarrollo de este gusano, incluso a nivel de células individuales y, además, tenían bibliotecas de mutantes, las cuales fueron críticas para revelar los futuros mecanismos regulatorios, incluidos el descubrimiento de los miRNAs (The New York Academy of Sciences, 2012).
En la década de los ochenta, la comunidad científica estaba concentrada únicamente en las proteínas denominadas factores de transcripción para describir los procesos de regulación genética y se ignoraban a la mayoría de las secuencias que no codificaban proteínas, a las cuales llamaban DNA basura. Por tanto, no había una base teórica que predijera que los sRNA pudieran regular la expresión genética.
El primer microRNA: lin-4
Los actores del descubrimiento del primer miRNA fueron dos investigadores que trabajaban en el laboratorio del Dr. Horvitz: Victor Ambros y Gary Ruvkun (Fig.3). Ambos estaban encargados de estudiar mutantes en genes heterocrónicos, es decir que estaban involucradas en la sincronización del desarrollo de C. elegans y dichas mutantes podían ser descritas fácilmente de forma visual (fenotípica) y funcionalmente (Almeida et al., 2011).

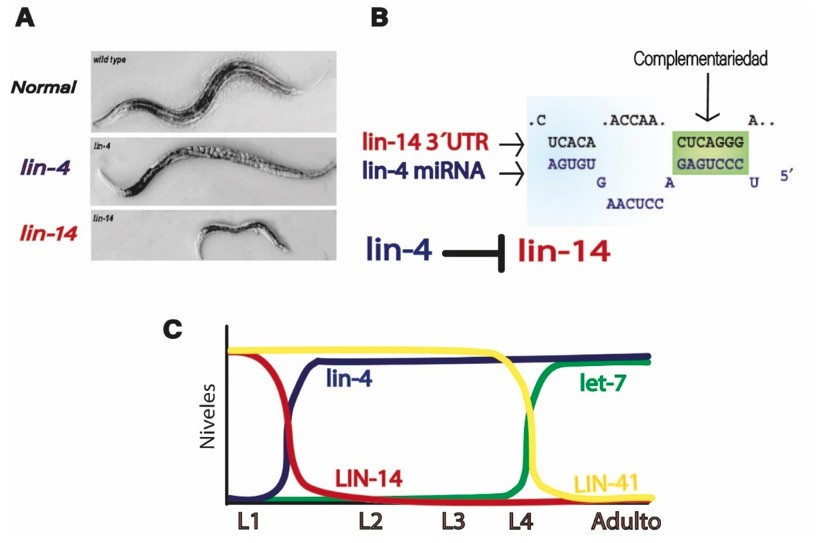
Ambros analizó la mutante del gen LIN-4, denominada lin-4, la cual había sido descubierta en los 70´s, y que causaba defectos severos en el desarrollo temporal de C. elegans. Esta mutante tenía un fenotipo retardado, en lugar de seguir un desarrollo normal, lin-4 repetía el primer estadio (L1) una y otra vez, perdiendo todas las partes que debería tener en etapas posteriores, en particular no desarrollaba vulva y, por tanto, la incapacidad de poner huevos. Por tanto, se concluyó que la actividad de LIN-4 era requerida para la transición de la etapa L1 a la L2 (Lee et al., 1993).
Al analizar y clonar el locus (posición del cromosoma donde se localiza una secuencia de DNA) que contenía a LIN-4, Victor Ambros junto con Rosalind Lee y Rhonda Feinbaum, encontraron un fragmento de 700 pares de bases (pb) que podría contener al gen LIN-4, pero no contenía los codones convencionales de inicio y de paro, los cuales eran comunes en genes que codificaban proteínas. Incluso introdujeron mutaciones en el supuesto marco de lectura abierto, sin embargo, la función de LIN-4 permanecía inalterable. Por tanto, Ambros concluyó que LIN-4 no codificaba a una proteína. Además, descubrieron que LIN-4 producía dos transcritos: uno de 61 nucleótidos que podía formar una estructura tipo tallo- asa y otro pequeño de 22 nts de longitud (Lee et al., 1993).
El blanco de lin-4: lin-14
Al mismo tiempo, Gary Ruvkun estaba enfocado en la mutante del gen LIN-14, lin-14, que tenía el fenotipo opuesto a la mutante lin-4. En lin-14, el nemátodo, en vez de quedarse en los primeros estadios larvales, no desarrollaba el primer estadio, sino que se saltaba hasta al segundo (L2). Estas mutantes eran muy pequeñas con un desarrollo precoz y desarrollaban algunas estructuras típicas del estadio adulto (Lee et al., 1993).
Además, de forma independiente, el grupo de investigación de Ruvkun encontró que LIN-14 codificaba a un factor de transcripción y que una región en el extremo 3´ del mRNA era suficiente para su regulación a nivel postranscripcional (Wightman et al., 1993).
De hecho, la mutación en LIN-14 era capaz de revertir el fenotipo de la mutación nula lin-4. Todos estos hallazgos hicieron pensar a los investigadores que LIN-4 podría regular negativamente a LIN-14, es decir, LIN-14 era el blanco de LIN-4. (Almeida et al., 2011). De esta forma, mientras que en la mutante lin-14 no existen los niveles de esta proteína necesarios para el desarrollo de la etapa L1, en la mutante lin-4, no se regula negativamente a lin-14, por eso los niveles de esta proteína se mantienen y no bajan en las etapas de L2 en adelante (Fig.4).
En junio de 1992, Ambros y Ruvnkun de forma independiente llegaron a la misma conclusión: los transcritos de LIN-4 eran complementarios a la secuencia repetida en la región 3´ no traducida (del inglés untranslated región, UTR) del gen LIN-14 (Lee et al., 1993, Wightman et al., 1993). Y en diciembre de 1993, ambos reportaron independientemente, en la misma emisión de la revista Cell, que el pequeño transcrito que no codifica a una proteína, lin-4, regula a LIN-14 través de su región UTR (Lee et al., 1993, Wightman et al., 1993) (Fig.4).
Es así que, un nuevo mecanismo celular regulatorio que involucra un transcrito no codificante había sido descrito. La complementariedad estaba conservada, así que la interacción de los dos debía ocurrir por apareamiento de bases. La conservación era un indicio de que debía existir una selección a través de la evolución debido a su importancia funcional. Al comparar los niveles de cambio del mRNA de LIN-14 con los de la proteína, se mostró que lin-4 está regulando la producción de esta proteína a un nivel de control traduccional.
A continuación, dos eventos posteriores fueron los que permitieron contextualizar y validar los resultados de Ambros y Ruvkun: el mecanismo del RNA interferente (RNAi) y el hallazgo del segundo miRNA. En 1998, Craig Mello y Andy Fire descubrieron el mecanismo de RNAi, dónde un RNA de doble hebra (dsRNA) exógeno modifica la actividad de un mRNA por complementariedad de secuencia (Fire et al. 1998).
El segundo miRNA: let-7
Ruvkun continuó explorando la genética de la vía heterocrónica y en el 2000 su grupo describió el segundo miRNA llamado let-7 que actuaba para llevar a cabo la transición del estadio L4 a adulto. En la mutante let-7 las células no terminaban de diferenciarse y continuaban dividiéndose hasta que el gusano moría (Reinhart et al, 2000). Con este hallazgo se pudo constatar que los miRNAs incrementaban en etapas específicas del desarrollo del nemátodo y reprimían blancos específicos que codifican para reguladores de desarrollo. En este caso el blanco de let-7 es LIN-41 (Reinhart et al., 2000) (Fig.4).
La relevancia de este nuevo miRNA es que, en contraste a lin-4, let-7 estaba conservado en un amplio rango de seres vivos, incluyendo el ser humano, mostrando que la regulación genética mediada por miRNAs es más antigua y común de lo que previamente se pensaba (The New York Academy of Sciences, 2012).
Funciones de los miRNAs en animales y plantas.
Los miRNAs pueden regular la expresión genética de eucariontes, de los cuales los miRNAs de animales y plantas son los mejores caracterizados. Esta regulación se lleva a cabo a diversos niveles, ya que, además de ser reguladores negativos de los transcritos, los miRNAs también pueden tener complementariedad con secuencias de promotores de genes favoreciendo o reprimiendo su transcripción y pueden, incluso, regular la biogénesis de otros miRNAs (Finnegan y Pasquinelli, 2013; O´Brien et al., 2018). En plantas, estos sRNAs tienen una complementariedad casi perfecta con su mRNA blanco, mientras que en animales solamente una región, denominada “región semilla” hibrida con su blanco (Ha and Kim, 2014).
Aunque el primer miRNA fue descubierto en C. elegans, actualmente se sabe que estos sRNAs están involucrados en prácticamente todos los procesos celulares necesarios para el desarrollo de los animales. Deleciones de los factores necesarios para la biogénesis de los miRNAs como DICER o DROSHA, causan letalidad en los embriones de ratón, además se ha determinado su función en el desarrollo de la mosquita de la fruta, ranas, pez cebra y en células de mamífero (Miska et al., 2005; Gebert y MacRae, 2019).
Enfocándonos en C. elegans, existen cerca de 140 genes que codifican para miRNAs en este nemátodo, los cuales participan en la vía heterocrónica, regulación del metabolismo, respuesta a cambios ambientales y en la longevidad (Ambros y Ruvkun, 2018). C. elegans es un modelo ampliamente usado en el área de ciencias biomédicas y biología del desarrollo porque comparte vías de señalización con el ser humano y puede ayudar a entender como es la regulación genética de diversas enfermedades humanas, ya que varios miRNAs son compartidos entre el nemátodo y el hombre (Lim et al., 2003). La expresión desregulada de los miRNA está asociada con el desarrollo de diversas enfermedades como las neurodegenerativas o autoinmunes e incluso han sido utilizados como biomarcadores durante el diagnóstico y pronóstico de cáncer. Por ejemplo, ahora se sabe que Let-7 es un importante supresor de tumores y tiene una expresión reducida en pacientes con cáncer de pulmón, asociándose a una menor supervivencia (Lee y Dutta, 2007; Almeida et al., 2011). En la tabla 1 se muestran algunos miRNAs compartidos por C. elegans y el ser humano, así como su función en diversos padecimientos.

En el caso de las plantas, los miRNAs han sido descritos como una nueva clase de moléculas de señalización que potencialmente se mueven célula-célula y actúan como señales de posición para establecer el plan corporal de estos organismos. Por ejemplo, los miRNAs miR165/miR166 regulan a la familia de factores de transcripción HD-ZIP III (HOMEODOMAIN-LEUCINE ZIPPER III) que están involucrados en el desarrollo del meristemo apical del brote, la parte apical del embrión y la polaridad de órganos laterales (Li & Zhang, 2006). El módulo miR396-GRF (GROWTH-REGULATING FACTORS), por otra parte, está implicado en la regulación del tamaño de la hoja y miR160 está involucrado en el desarrollo de la raíz primaria, el incremento de raíces laterales y el desarrollo de caliptra (la cubierta que cubre la punta de la raíz) a través de la regulación de sus blancos los Factores de Respuesta a Auxinas: ARF10/16 (Wang et al., 2005). Además, miR172 y miR156 están implicados en la transición de la fase vegetativa a la reproductiva, la sobreexpresión de miR156 y la reducción de su blanco SQUAMOSA-PROMOTOR BINDING PROTEIN-LIKE (SPL) retrasa la floración, mientras que la sobreexpresión de miR172 la promueve. De esta forma, mientras miR156 especifica la fase juvenil, miR172 determina la fase reproductiva degradando a sus blancos, los transcritos APETALA2-LIKE (AP2-LIKE) (D´Ario et al., 2017) (Figura 5).
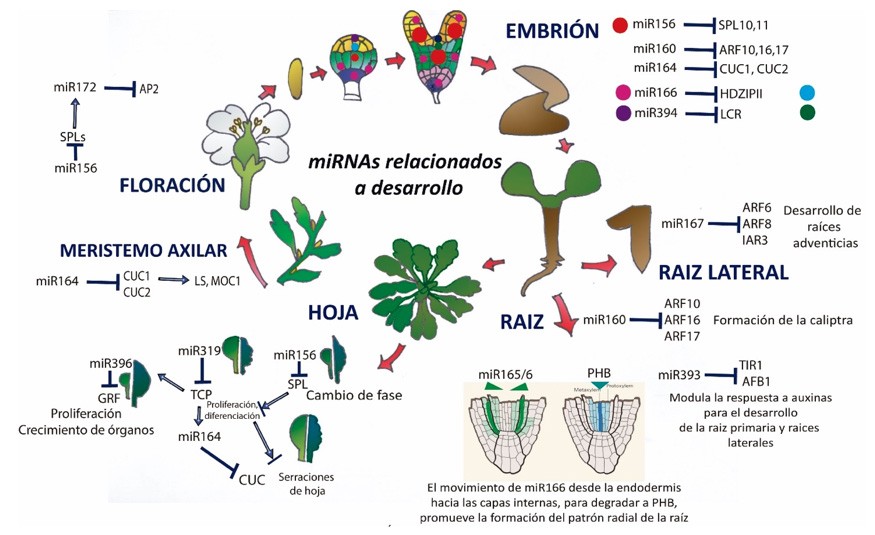
A parte de los miRNAs conservados, descritos arriba, que pueden regular características comunes en las plantas, como su morfología y cambio de fase, existen miRNAs que son especie-específicos y pueden controlar procesos únicos en las plantas como el desarrollo de las fibras en el algodón (Zhang, 2015). Ambos tipos de miRNAs juegan un rol importante en respuesta a factores abióticos y se han propuesto como blancos para mejorar a los cultivos e incrementar su tolerancia a diferentes tipos de estrés, tal como se muestra en la Tabla 2.

En conclusión, los miRNAs descubiertos por Ambros y Ruvkun son parte de una larga clase de sRNAs que modifican la expresión de genes y tienen una amplia función en el desarrollo de diversos organismos. Este mecanismo mediado por RNA contrastaba con el modelo universal de regulación genética impuesto hasta entonces, donde los factores de transcripción se unen al DNA y modifican la transcripción de un gen. Se comenzó así, a poner más atención a los RNA pequeños como moléculas muy importantes en la regulación genética y con un enorme potencial como biomarcadores en diversos padecimientos en seres humanos, así como una importante herramienta biotecnológica para el mejoramiento de los cultivos.
Referencias
Almeida M., R. Reis and G. Calin. 2011. MicroRNA history: Discovery, recent applications and next frontiers. Mutation research 717:1-8.
Ambros, V., & Ruvkun, G. (2018). Recent molecular genetic explorations of Caenorhabditis elegans microRNAs. Genetics, 209(3), 651-673.
Balzeau, J., Menezes, M. R., Cao, S., & Hagan, J. P. (2017). The LIN28/let-7 pathway in cancer. Frontiers in genetics, 8, 31.
Boyerinas, B., Park, S. M., Hau, A., Murmann, A. E., & Peter, M. E. (2010). The role of let-7 in cell differentiation and cancer. Endocrine-related cancer, 17(1), F19-F36.
D’Ario, M., Griffiths-Jones, S., & Kim, M. (2017). Small RNAs: big impact on plant development. Trends in Plant Science, 22(12), 1056-1068.7
Finnegan, E. F., & Pasquinelli, A. E. (2013). MicroRNA biogenesis: regulating the regulators. Critical reviews in biochemistry and molecular biology, 48(1), 51-68.
Fire, A. S. Xu, M. Montgomery, S. Kostas, S. Driver and C. Mello et al. 1998. Potent and specific genetic interference by double stranded RNA in Caenorhabditis elegans. Nature 391: 806–811.
Gebert, L. F., & MacRae, I. J. (2019). Regulation of microRNA function in animals. Nature reviews Molecular cell biology, 20(1), 21-37.
Ha, M., & Kim, V. N. (2014). Regulation of microRNA biogenesis. Nature reviews Molecular cell biology, 15(8), 509-524.
He, W., Huang, Y., chuan Jiang, C., Zhu, Y., Wang, L., Zhang, W., … & Tang, S. (2020). miR-100 inhibits cell growth and proliferation by targeting HOXA1 in nasopharyngeal carcinoma. OncoTargets and therapy, 13, 593.
Kura, B., Kalocayova, B., Devaux, Y., & Bartekova, M. (2020). Potential clinical implications of miR-1 and miR-21 in heart disease and cardioprotection. International journal of molecular sciences, 21(3), 700.
Lee R, R. Feinbaum and V. Ambros. 2004. A short history of a short RNA. Cell 116: S89–S92.
Lee R. R. Feinbaum, V. Ambros. 1993. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 75 (5): 843–854.
Li, C., & Zhang, B. (2016). MicroRNAs in control of plant development. Journal of Cellular Physiology, 231(2), 303-313.
Lim, L. P., Lau, N. C., Weinstein, E. G., Abdelhakim, A., Yekta, S., Rhoades, M. W., … & Bartel, D. P. (2003). The microRNAs of Caenorhabditis elegans. Genes & development, 17(8), 991-1008.
Liu, R., Wang, M., Li, E., Yang, Y., Li, J., Chen, S., … & Hu, Z. (2020). Dysregulation of microRNA-125a contributes to obesity-associated insulin resistance and dysregulates lipid metabolism in mice. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 1865(5), 158640.
López Ruiz Brenda Anabel. 2019. Tesis que para obtener el grado de Doctora en Ciencias Bioquímicas. Departamento de Bioquímica, UNAM. Página 31.
Madelaine, R., Sloan, S. A., Huber, N., Notwell, J. H., Leung, L. C., Skariah, G., … & Mourrain, P. (2017). MicroRNA-9 couples brain neurogenesis and angiogenesis. Cell reports, 20(7), 1533-1542.
Miska, E. A. (2005). How microRNAs control cell division, differentiation and death. Current opinion in genetics & development, 15(5), 563-568.
Nair, P. (2011). Profile of Gary Ruvkun. Proceedings of the National Academy of Sciences, 108(37), 15043-15045.
O’Brien, J., Hayder, H., Zayed, Y., & Peng, C. (2018). Overview of microRNA biogenesis, mechanisms of actions, and circulation. Frontiers in endocrinology, 9, 402.
Reinhart B., F. Slack, M. Basson, A. Pasquinelli, J. Bettinger, A. Rougvie, H. Horvitz, G. Ruvkun. 2000. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature 403 (6772): 901–906.
The New York Academy of Sciences. 2012. The microRNA Revolution: the 2012 Dr. Paul Janssen Award Symposium. Meeting Report. Disponible en: https://www.nyas.org/ebriefings/the-microrna-revolution/?tab=the%20discovery%20of%20the%20first%20micrornas
Wang, J. W., Wang, L. J., Mao, Y. B., Cai, W. J., Xue, H. W., & Chen, X. Y. (2005). Control of root cap formation by microRNAtargeted auxin response factors in Arabidopsis. The Plant Cell, 17(8), 2204-2216
Wani, S. H., Kumar, V., Khare, T., Tripathi, P., Shah, T., Ramakrishna, C., … & Mangrauthia, S. K. (2020). miRNA applications for engineering abiotic stress tolerance in plants. Biologia, 75(7), 1063-1081.
Wightman B., I. Ha and G. Ruvkun. 1993. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell 75 (5). 855–862.
www.wormatlas.org/ver1/handbook/anatomyintro/anatomyintro.html. Fecha de consulta: 27 de enero de 2017.
Zhang, B. (2015). MicroRNA: a new target for improving plant tolerance to abiotic stress. Journal of experimental botany, 66(7), 1749-1761.
Zhang, B., & Wang, Q. (2015). MicroRNA‐based biotechnology for plant improvement. Journal of cellular physiology, 230(1), 1-15.