En la actualidad podemos inferir la evolución de las especies a partir de las diferencias entre sus genes y proteínas; el estudio de estas diferencias también permite identificar los aminoácidos relevantes para la función enzimática. En este artículo analizamos cómo se puede reconstruir la historia evolutiva de las moscas de la fruta, empleando una enzima que degrada alcohol, a la vez que identificamos el sitio activo de esta proteína.
La evolución arborescente
Gracias al trabajo de Charles Darwin se sabe que las especies evolucionamos por divergencia a partir de ancestros comunes. De esta forma, los humanos y chimpancés nos transformamos a partir de una especie ancestral que existió hace aproximadamente 6 millones de años; la que a su vez, comparte un ancestro común con los gorilas hace 9 millones de años. Este patrón de evolución se puede describir como un árbol en donde las especies actuales son las hojas y los ancestros comunes, los nodos de las ramas. De hecho, esta teoría es tan importante que cada 12 de febrero se celebra el Día de Darwin a nivel mundial. En El origen de las especies (1859) Darwin escribe:
“Las afinidades de todos los seres de la misma clase se han representado algunas veces por un gran árbol.”
Y la única figura que contiene El origen de las especies ilustra precisamente este proceso de evolución por divergencia (Figura 1).

Es importante recalcar que con esta figura Darwin también pretendía ejemplificar el principio de la ventaja derivada de la divergencia de caracteres. Según esto, las variedades que divergen más, tenderán a conservarse y acumularse por selección natural a lo largo de las generaciones. Darwin explica cómo a partir de la especie ancestral A evolucionan dos variedades (a1 y m1) que a su vez dan origen a las variedades a2, s2 y m2. Éstas se distinguirán de la especie ancestral A más de lo que difirieron a1 y m1. El resultado es que después de catorce mil generaciones (cada línea horizontal representa mil generaciones), la especie A origina ocho especies nuevas. Un proceso similar ocurre con la especie ancestral I. Por supuesto, este proceso no es constante ni uniforme y algunas especies se extinguen en el camino.
Las moléculas como documentos históricos
A mediados de los años 60’s Emile Zuckerkandl y Linus Pauling publican una serie de artículos en donde sugieren que el DNA, RNA y las proteínas contienen información que puede utilizarse para reconstruir la historia evolutiva de los seres vivos. También proponen la hipótesis del reloj molecular, la cual establece que dichas moléculas acumulan cambios a una tasa aproximadamente constante a lo largo del tiempo. Su trabajo fue pionero en la disciplina de la evolución molecular.
El artículo en donde Emile Zuckerckandl y Linus Pauling plantean la hipótesis del reloj molecular no estuvo sometido a revisión por pares. Es decir, no fue revisado por otros científicos antes de su publicación, sino que fue el resultado de una invitación hecha a Linus Pauling para difundir un artículo como parte de un volumen especial (Festschrift) para celebrar al bioquímico descubridor de la vitamina C y Premio Nobel de Medicina Albert Szent-Györgyi. El laureado opinaba que los científicos se podían dividir en apolíneos o dionisíacos. Los apolíneos tienden a desarrollar líneas de investigación ya establecidas hasta la perfección; en tanto que los dionisíacos confían más en su intuición y se inclinan por realizar descubrimientos inesperados.
En sintonía con la aproximación dionisíaca a la ciencia, Linus Pauling le pidió a Emile Zuckerkandl, quien por aquellos años trabajaba en su laboratorio, que escribiera “¡un artículo escandaloso!” dado que se trataba de un homenaje a Albert Szent-Györgyi. De esa forma publicaron “Molecular Disease, Evolution and Genic Heterogeneity” en 1962. Este artículo, junto con otros más, publicados por Linus Pauling y Emile Zuckerkandl incitaron a los biólogos a estudiar la evolución de los seres vivos a partir de sus genes.
La enzima alcohol deshidrogenasa y las moscas de la fruta
La mosca amante de la fruta de vientre negro, Drosophila melanogaster es un organismo modelo que en biología se ha empleado para realizar importantes descubrimientos. Por ejemplo, a principios del siglo XX Thomas Morgan propuso que los genes residen en los cromosomas estudiando la genética de estos insectos.
En 1983 Marty Kreitman publicó un artículo en donde describía por primera vez la variación genética a nivel del DNA en la naturaleza. Para ello, secuenció el gen codificante de la enzima alcohol deshidrogenasa de 11 moscas silvestres. El trabajo requirió un esfuerzo colosal dadas las tecnologías de la época.
La alcohol deshidrogenasa es una enzima que cataliza la interconversión entre alcoholes y aldehídos (o cetonas) con la concomitante reducción de NAD+ a NADH:
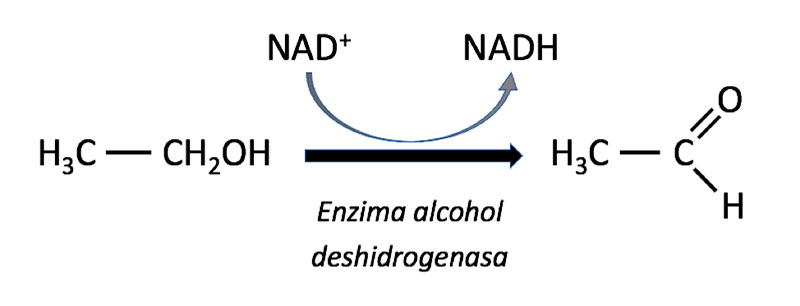
Las Drosophila contienen esta enzima en abundancia. Su presencia les permite tolerar los altos niveles de alcohol que encuentran en la fruta en descomposición. Esta enzima ha sido ampliamente estudiada en Drosophila para tratar de entender la relación que existe entre la variación genética y la adaptación.
Para estudiar la evolución de la alcohol deshidrogenasa es necesario comparar varias enzimas. Por fortuna, las bases de datos genéticas están repletas de homólogos de esta enzima; mismas que comparten un ancestro común. Estas bases de datos son de acceso gratuito gracias al trabajo pionero de Margaret Dayhoff, científica que argumentaba que todo mundo debería tener acceso a la información de las secuencias de proteínas, dado que se habían generado con dinero público. Para encontrar enzimas homólogas, basta realizar una simple búsqueda en Genbank con una herramienta computacional que se llama BLAST.
Estas enzimas se pueden alinear siguiendo su estructura primaria de aminoácidos. El objetivo de hacer una alineación múltiple de proteínas es la identificación de los aminoácidos homólogos entre las distintas enzimas. En cada renglón se coloca una proteína y los aminoácidos que son homólogos, se alinean en una misma columna.
Al igual que en la película de Matrix en donde los personajes podían obtener información leyendo la lluvia digital, los biólogos podemos obtener la información que contiene una alineación múltiple y utilizarla para inferir la historia evolutiva de las enzimas y especies (Figura 2).

El patrón que se observa en la alineación múltiple de esta enzima es el resultado de aproximadamente 43 millones de años de evolución. Si el tiempo que tarda una Drosophila en convertirse en adulto reproductivo es de 10 días, y asumimos que este tiempo se ha mantenido constante a lo largo de la evolución, entonces hay cerca de un millón de generaciones de moscas entre el ancestro común y cada una de las secuencias de aminoácidos actuales. En este tiempo, un sinfín de historias-mosca han ocurrido en donde aparecieron variantes genéticas que contribuyeron a la supervivencia diferencial de individuos.
A partir de contar las diferencias entre las secuencias de aminoácidos se puede reconstruir la filogenia de las enzimas (Figura 3), que es una aproximación de la historia evolutiva de las especies del género Drosophila. Seguramente Darwin estaría sorprendido de ver cómo sus ideas cristalizaron en técnicas concretas para inferir la evolución y divergencia de las especies a partir de un ancestro común.
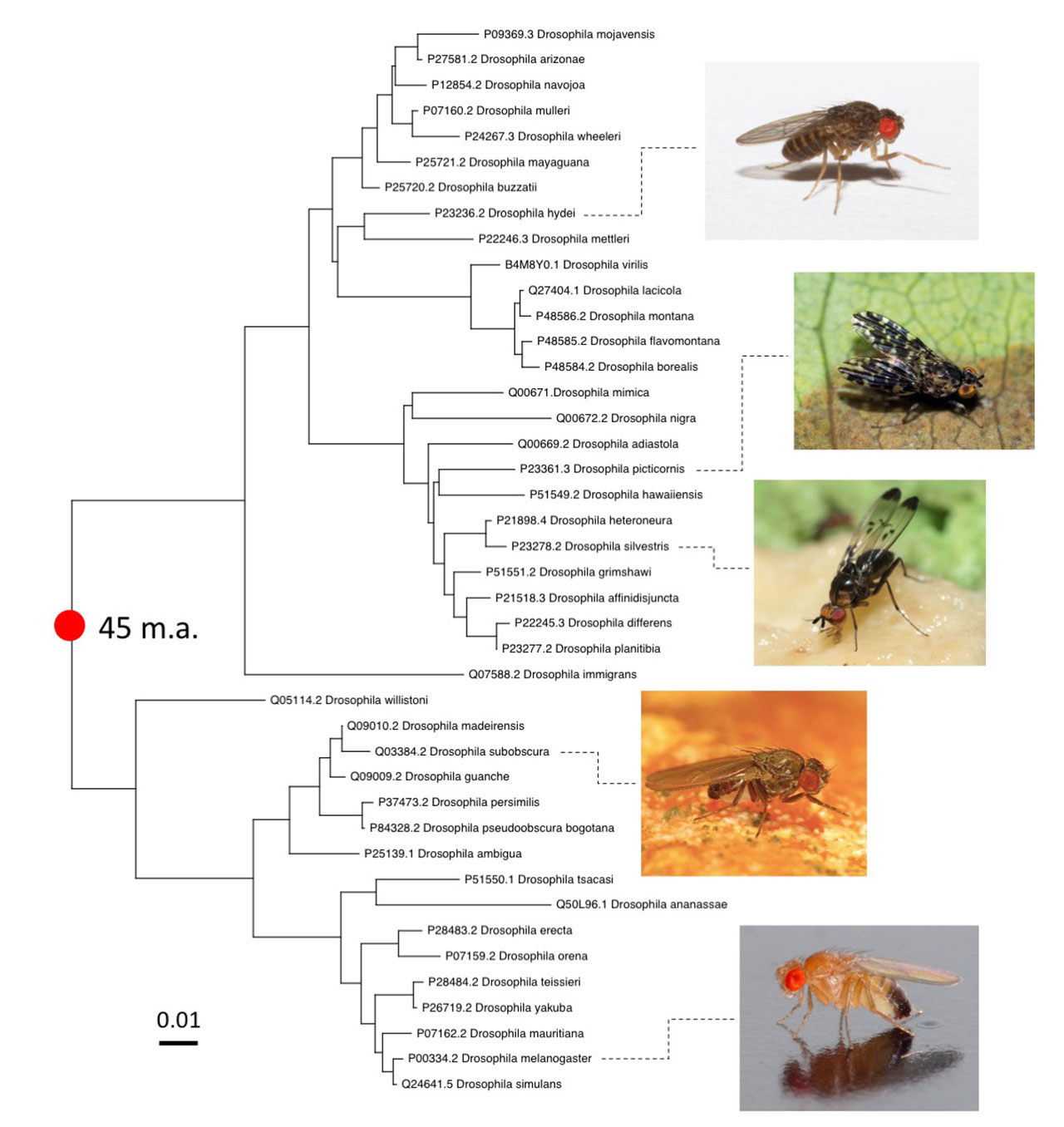
(By Martin Cooper – https://www.flickr.com/photos/m-a-r-t-i-n/23734393432/, CC BY 2.0, https://commons.wikimedia.org/w/index.php?curid=83282911); Drosophila silvestris
(By KarlM (talk) – I (KarlM (talk)) created this work entirely by myself., CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=99258244); Drosophila picticornis
(By Shchurch – Own work, CC BY-SA 4.0, https://commons.wikimedia.org/w/index.php?curid=118332140); Drosophila hydei
(By Brian Gratwicke – Flickr: Drosophila hydei larger fruitflies for small frogs, CC BY 2.0, https://commons.wikimedia.org/w/index.php?curid=12995685)
Las mutaciones y la selección natural
Las diferencias que se observan entre las enzimas pueden deberse a la selección natural o a la deriva genética. Una vez que una mutación ocurre, su destino depende en gran medida de su impacto en el individuo. Los efectos de la mutación pueden ser negativos, neutrales o positivos.
La mayoría de las mutaciones parecen tener un efecto negativo y normalmente no se transmiten a las generaciones subsecuentes porque causan un efecto severo en los individuos en los que ocurren. Otro porcentaje importante de las mutaciones que ocurren tienen un efecto ligeramente negativo o neutral. Se piensa que la mayoría de las diferencias observadas entre las proteínas tienen este efecto. Las mutaciones con efecto positivo son las menos comunes.
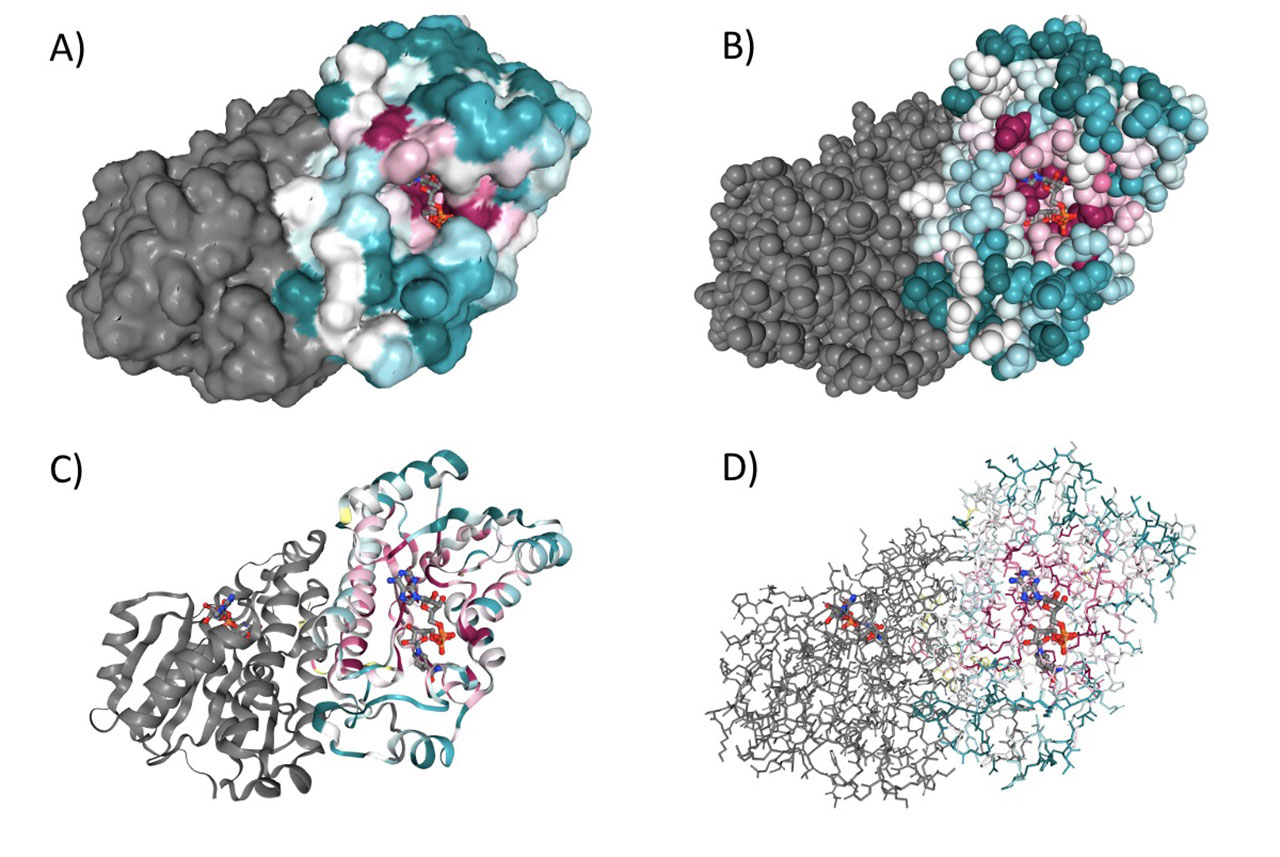
El efecto de las mutaciones en el funcionamiento de las enzimas no se distribuye de manera aleatoria a lo largo de su estructura terciaria. Por ejemplo, si una de ellas ocurre en una región del DNA que cambia un aminoácido de la superficie de la enzima, hay más probabilidades que tenga un efecto levemente deletéreo o neutral en la función de la enzima y por lo tanto se pueda heredar a las generaciones subsecuentes. Por otro lado, si la mutación cambia un aminoácido directamente comprometido con la función enzimática, es más factible que tenga un efecto deletéreo y no se herede a la siguiente generación.
Si coloreamos los aminoácidos de la enzima alcohol deshidrogenasa de D. melanogaster de acuerdo con su nivel de variación, veremos que los aminoácidos que más cambian entre las especies son los que tienden a estar en la superficie de la proteína, en tanto que los más conservados propenden a localizarse hacia el interior. En particular, los aminoácidos que se encuentran en el sitio activo son los más conservados de todos (Figura 4).
Conclusiones
El estudio de la evolución molecular tiene sus raíces en la teoría de evolución de Darwin. La propuesta de utilizar a los genes y proteínas como documentos históricos para inferir la evolución de los seres vivos se debe al trabajo creativo de Emile Zuckerckandl y Linus Pauling. Utilizando a la enzima alcohol deshidrogenasa como marcador evolutivo, puede inferirse la evolución de distintas especies del género Drosophila. La filogenia que se obtiene nos recuerda a la historia evolutiva que Darwin imaginó y dibujó para su libro El origen de las especies. Finalmente, al estudiar las diferencias que se observan entre las enzimas de distintas especies, se pueden identificar aminoácidos relevantes para la función enzimática. Dichos aminoácidos tienden a mostrar en dónde está el sitio activo de la enzima. La teoría de la evolución permite entender el patrón de variación que se encuentra entre los genes y las proteínas de distintas especies.
Para saber más:
Szent-Györgyi A. Dionysians and apollonians. Science. 1972 Jun 2;176(4038):966.
https://www.science.org/doi/10.1126/science.176.4038.966.a
Gregory J. Morgan. 1998. Emile Zuckerkandl, Linus Pauling, and the Molecular Evolutionary Clock, 1959-1965. Journal of the History of Biology. 31: 155-178.
Suárez Díaz, Edna. (2017) Evolución y Moléculas: la molecularización de la biología evolutiva en contexto. Ciudad de México, México: Universidad Nacional Autónoma de México y Centro de Estudios Filosóficos, Políticos y Sociales Vicente Lombardo Toledano.