El cuerpo humano está constituido por más de 30 billones de células que conforman los diferentes órganos y tejidos, con funciones determinadas para cada uno (Grossman et al., 2016). A pesar de que todas las células que constituyen el cuerpo humano tienen el mismo material genético o ADN, no todas expresan los mismos genes, debido a diversos mecanismos moleculares que se encargan de regular la expresión de genes de manera específica, lo que conduce a que se sinteticen únicamente las proteínas necesarias para que cada tipo de célula pueda llevar a cabo su función. Uno de estos mecanismos incluye la inhibición de la expresión de genes mediada por ARNs no codificantes, denominados así por porque no se traducen a proteínas (Catalanotto et al., 2016). Dentro de los ARNs no codificantes, se encuentra un grupo conocido como microARNs (miARNs), porque tiene un tamaño de 19-25 nucleótidos e inhibe la expresión de sus genes blancos para promover la degradación de sus ARN mensajeros (ARNm) blancos o inhibir la traducción de estos (Oliveto et al., 2017).
En humanos se han identificado aproximadamente 2,200 miARNs, que regulan la expresión de más del 60% de todos los genes que codifican para proteínas, por lo que alteraciones en la expresión de los miARNs promueven el desarrollo de enfermedades, incluyendo el cáncer (Tian et al., 2022). Los miARNs pueden secretarse hacia el exterior de las células (conocidos como miARNs circulantes o exomiARNs) y están presentes en prácticamente todos los fluidos biológicos, incluyendo líquido cefalorraquídeo, saliva, suero, plasma y orina, siendo moléculas de fácil obtención (Condrat et al., 2020). Los miARNs también pueden ser secretados a través de vesículas por las células cancerosas hacia el torrente sanguíneo, lo que les permite llegar a otros tejidos u órganos. Estudios previos han encontrado que el 10% de los miRNAs circulantes son secretados en exosomas y que el 90% restante forma complejos con proteínas como Argonauta 2 (Ago2), Nucleosplamina 1 (NPM 1) y lipoproteínas de alta densidad (HPL). Esto es importante pues previene la degradación de los miARNs por las ARNasas presentes en los fluidos corporales (Alotaibi, 2023). Además, estos miARNs son muy específicos para cada tipo de cáncer, por lo que constituyen firmas moleculares cáncer-especificas. Otra propiedad considerable de estos miARNs es que se pueden detectar a partir de cantidades suficientes por RT-qPCR y que están presentes desde las etapas tempranas hasta las tardías del cáncer (Alotaibi, 2023), lo que los convierte en moléculas ideales como biomarcadores de diagnóstico y pronóstico temprano en diferentes tipos de cáncer (Condrat et al., 2020).
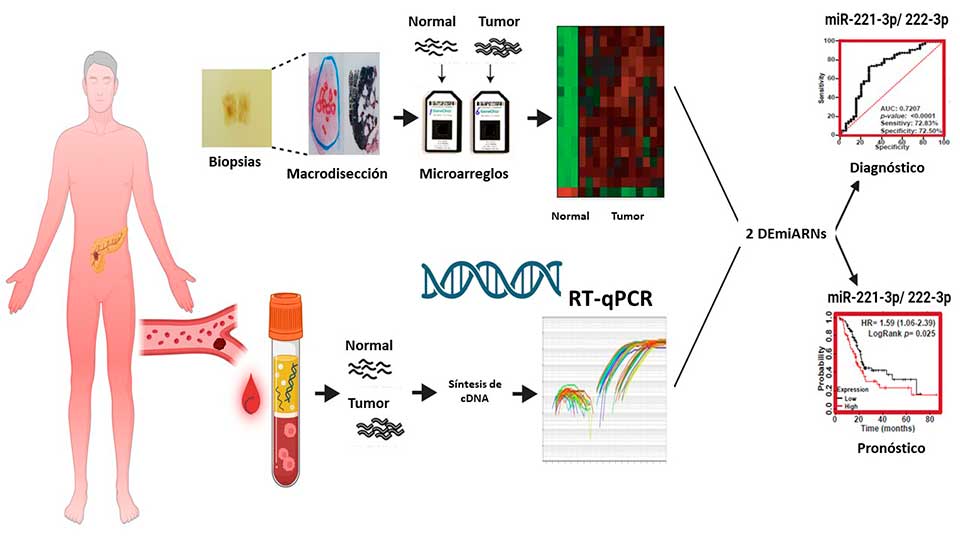
En 2022 se reportaron 19.976 millones de nuevos casos de cáncer, siendo el Adenocarcinoma Ductal Pancreático (ACDP) uno de los cánceres más letales, por lo que ocupa el 6° lugar en mortalidad en el mundo con un índice de fatalidad del 0.96 (Ferlay et al., 2024). Ello se debe a que el diagnóstico temprano del ACDP es difícil debido a la ausencia de síntomas específicos, así como al tamaño pequeño de los tumores (<1 cm) por lo que no pueden ser detectados por pruebas de imagenología, y aunque el antígeno CA 19-9 (en suero) ha sido propuesto como una herramienta de diagnóstico, tiene baja sensibilidad (70-90%) y especificidad (68-91%), además de que se han observado niveles elevados de este antígeno en condiciones benignas, como la colangitis, por lo que la biopsia sigue siendo el estándar de oro para el diagnóstico de ACDP, a pesar de que es un método invasivo, costoso y requiere hospitalización (Wood et al., 2022), por lo que es imperativa la búsqueda de nuevos biomarcadores de diagnóstico que tengan una alta sensibilidad, especificidad y que no sean invasivos para el diagnóstico del ACDP. Los miARNs han surgido como moléculas prominentes para el diagnóstico de esta enfermedad, de la cual ya han sido identificados varios miARNs en el suero o plasma de pacientes (Mok et al., 2024). Sin embargo, si consideramos que la heterogeneidad genética en cada población afecta la expresión y función de los miARNs en individuos con diferentes orígenes étnicos (Flowers et al., 2022), se hace necesario conocer el perfil de miARNs de cada población en cada tipo de cáncer. Considerando que en México el ACDP ocupa el 12avo lugar en incidencia y el 7mo en mortalidad (Ferlay et al., 2024) y que hasta la fecha no se han identificado los miARNs presentes en pacientes con ACDP que residen en México, es que, en este estudio (Álvarez-Hilario et al., 2023), identificamos miARNs diferencialmente expresados (DEmiARNs) en plasma de pacientes con ACDP que residen en México y se compararon contra el plasma de individuos control para identificar una firma de miARNs en pacientes mexicanos que pudiera usarse en el diagnóstico y pronóstico de esta enfermedad (Figura 1).
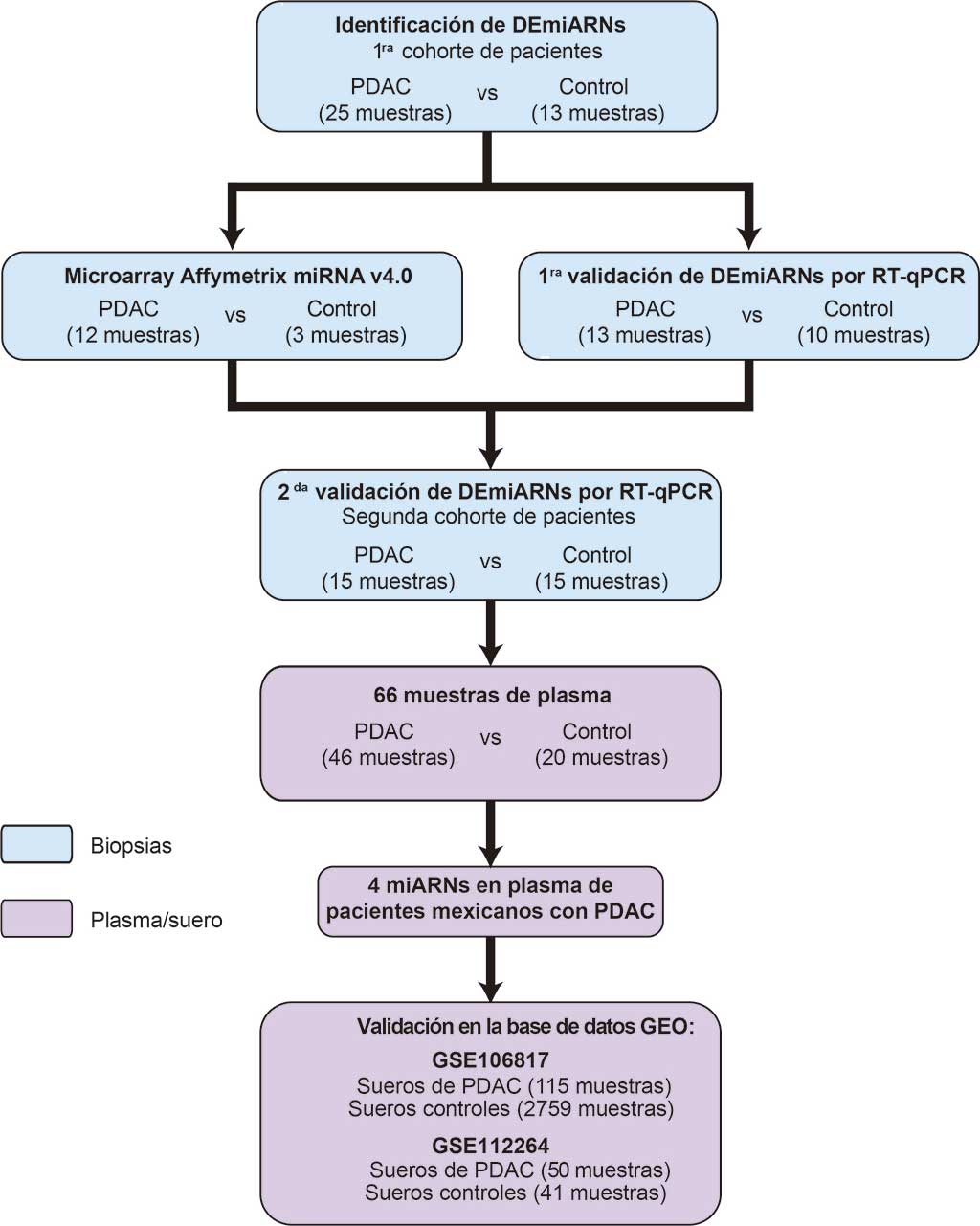
Inicialmente y debido a que no se conocía el miRNoma del ACDP en pacientes que residen en México se procedió a averiguar los DEmiRNAs que estaban presentes en biopsias de pacientes con ACDP, pero no en biopsias de individuos control, para después identificar a estos DEmiARNs exclusivos en individuos con ACDP en plasma. Sin embargo, antes de recuperar los miARNs de ambas muestras, confirmamos la presencia de células tumorales en las biopsias de ACDP y su ausencia en las biopsias control mediante inmunohistoquímica. A partir de esto, las células tumorales se aislaron de las biopsias mediante macrodisección y se extrajo el RNA, identificando 17 DEmiARNs a través de microarreglos. Éstos fueron validados en 2 cohortes independientes de muestras de biopsias de pacientes con ACDP y de individuos control, encontrando que 10 de los 17 DEmiARNs constituyen una firma para estos pacientes en México. De estos 10 DEmiRNAs identificados en biopsias, solo 4 DEmiARNs (miR-222-3p, miR-345-5p, miR-100-5p y miR-221-3p) se hallaron en muestras de plasma de pacientes con ACDP mediante RT-qPCR. Dos de los cuales (miR-221.3p y miR-222.3p), mediante el análisis de ROC y las curvas de Kaplan-Meier, se demostró que pueden ser empleados en el diagnóstico y pronóstico del ACDP en personas que residen en la República Mexicana.
Finalmente, para identificar los posibles genes blancos de estos 4 DEmiARNs y su papel en ACDP, se realizó un análisis bioinformático, encontrando 47 genes blancos potenciales; los cuales participan en vías metabólicas como la oxidación de ácidos grasos, la homeostasis de la glucosa, los procesos catabólicos de compuestos nitrogenados y el transporte de aminoácidos, todos ellos procesos metabólicos que participan en la reprogramación metabólica y la patobiología del ACDP, una de las marcas de las células cancerosas (Faubert et al., 2020).
En conclusión, en este trabajo se identificaron al menos dos miARNs (miR-222-3p y miR-221-3p), que podrían ser usados como biomarcadores no invasivos, sensibles y específicos para el diagnóstico y pronóstico temprano de esta enfermedad en pacientes con ACDP que residen en México (Figura 2) y se estableció por primera vez el miRNoma del ACDP de pacientes mexicanos (Álvarez-Hilario et al., 2023).
Referencias
Alotaibi, F. (2023). Exosomal microRNAs in cancer: Potential biomarkers and immunotherapeutic targets for immune checkpoint molecules. Front Genet, 14, 1052731. https://doi.org/10.3389/fgene.2023.1052731
Álvarez-Hilario, L. G., Salmeron-Barcenas, E. G., et al (2023). Circulating miRNAs as Noninvasive Biomarkers for PDAC Diagnosis and Prognosis in Mexico. Int J Mol Sci, 24(20). https://doi.org/10.3390/ijms242015193
Catalanotto, C., Cogoni, C., & Zardo, G. (2016). MicroRNA in Control of Gene Expression: An Overview of Nuclear Functions. Int J Mol Sci, 17(10). https://doi.org/10.3390/ijms17101712
Condrat, C. E., Thompson, D. C., Barbu, et al (2020). miRNAs as Biomarkers in Disease: Latest Findings Regarding Their Role in Diagnosis and Prognosis. Cells, 9(2). https://doi.org/10.3390/cells9020276
Faubert, B., Solmonson, A., & DeBerardinis, R. J. (2020). Metabolic reprogramming and cancer progression. 368(6487), eaaw5473. https://doi.org/doi:10.1126/science.aaw5473
Ferlay, J., Ervik M, Lam F, et al (2024). Global Cancer Observatory: Cancer Today. Lyon, France: International Agency for Research on Cancer. Int J Cancer. https://gco.iarc.fr/en
Flowers, E., Kanaya, A. M., Zhang, L., & Aouizerat, B. E. (2022). The Role of Racial and Ethnic Factors in MicroRNA Expression and Risk for Type 2 Diabetes [Brief Research Report]. 13. https://doi.org/10.3389/fgene.2022.853633
Grossman, R. L., Heath, A. P., Ferretti, V., et al (2016). Toward a Shared Vision for Cancer Genomic Data. N Engl J Med,375(12), 1109-1112. https://doi.org/10.1056/NEJMp1607591
Mok, E. T. Y., Chitty, J. L., & Cox, T. R. (2024). miRNAs in pancreatic cancer progression and metastasis. Clin Exp Metastasis. https://doi.org/10.1007/s10585-023-10256-0
Oliveto, S., Mancino, M., Manfrini, N., & Biffo, S. (2017). Role of microRNAs in translation regulation and cancer. World J Biol Chem, 8(1), 45-56. https://doi.org/10.4331/wjbc.v8.i1.45
Tian, S., Wang, J., Zhang, F., & Wang, D. (2022). Comparative Analysis of microRNA Binding Site Distribution and microRNA-Mediated Gene Expression Repression of Oncogenes and Tumor Suppressor Genes. Genes (Basel),13(3). https://doi.org/10.3390/genes13030481
Foto de portada: Micro RNA (miRNA) Ilustración 3d